+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4tw1 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of the octameric pore complex of the Staphylococcus aureus Bi-component Toxin LukGH | ||||||
 要素 要素 | (Possible leukocidin subunit) x 2 | ||||||
 キーワード キーワード | TOXIN / octamer leukocidin pore-forming toxin | ||||||
| 機能・相同性 | Leukocidin/porin MspA / Leukocidin-like / Distorted Sandwich / Mainly Beta / : / :  機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.8 Å 分子置換 / 解像度: 2.8 Å | ||||||
 データ登録者 データ登録者 | Logan, D.T. / Hakansson, M. / Saline, M. / Kimbung, R. / Badarau, A. / Rouha, H. / Nagy, E. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2015 ジャーナル: J.Biol.Chem. / 年: 2015タイトル: Structure-Function Analysis of Heterodimer Formation, Oligomerization, and Receptor Binding of the Staphylococcus aureus Bi-component Toxin LukGH. 著者: Badarau, A. / Rouha, H. / Malafa, S. / Logan, D.T. / Hakansson, M. / Stulik, L. / Dolezilkova, I. / Teubenbacher, A. / Gross, K. / Maierhofer, B. / Weber, S. / Jagerhofer, M. / Hoffman, D. / Nagy, E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4tw1.cif.gz 4tw1.cif.gz | 910.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4tw1.ent.gz pdb4tw1.ent.gz | 753.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4tw1.json.gz 4tw1.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4tw1_validation.pdf.gz 4tw1_validation.pdf.gz | 585.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4tw1_full_validation.pdf.gz 4tw1_full_validation.pdf.gz | 720.5 KB | 表示 | |
| XML形式データ |  4tw1_validation.xml.gz 4tw1_validation.xml.gz | 164.4 KB | 表示 | |
| CIF形式データ |  4tw1_validation.cif.gz 4tw1_validation.cif.gz | 223.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/tw/4tw1 https://data.pdbj.org/pub/pdb/validation_reports/tw/4tw1 ftp://data.pdbj.org/pub/pdb/validation_reports/tw/4tw1 ftp://data.pdbj.org/pub/pdb/validation_reports/tw/4tw1 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 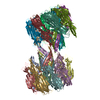
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質 | 分子量: 35825.539 Da / 分子数: 8 / 由来タイプ: 組換発現 由来: (組換発現)  株: USA300 / TCH1516 / 遺伝子: USA300HOU_2011 / 発現宿主:  #2: タンパク質 | 分子量: 37682.680 Da / 分子数: 8 / Fragment: Residues 28-351 / 由来タイプ: 組換発現 由来: (組換発現)  株: USA300 / TCH1516 / 遺伝子: USA300HOU_2013 / 発現宿主:  #3: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 4.39 Å3/Da / 溶媒含有率: 71.96 % / 解説: Thin plates |
|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.5 詳細: Drops consisted of 100 nl protein in 20 mM HEPES buffer pH 7.5 and 100 nl reservoir 30 mM MgCl2, 30 mM CaCl2, 0.1 M Na HEPES/MOPS buffer pH 7.5, 12.5 % v/v MPD, 12.5 % w/v PEG 1000, 12.5 % ...詳細: Drops consisted of 100 nl protein in 20 mM HEPES buffer pH 7.5 and 100 nl reservoir 30 mM MgCl2, 30 mM CaCl2, 0.1 M Na HEPES/MOPS buffer pH 7.5, 12.5 % v/v MPD, 12.5 % w/v PEG 1000, 12.5 % w/v PEG 3350. Crystals appeared after 7 days. |
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  MAX II MAX II  / ビームライン: I911-3 / 波長: 1 Å / ビームライン: I911-3 / 波長: 1 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 検出器 | タイプ: MARMOSAIC 225 mm CCD / 検出器: CCD / 日付: 2013年12月12日 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射 | モノクロメーター: Double crystal / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 放射波長 | 波長: 1 Å / 相対比: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 | 解像度: 2.8→48.188 Å / Num. all: 222018 / Num. obs: 222018 / % possible obs: 97.9 % / 冗長度: 3.1 % / Biso Wilson estimate: 56.4 Å2 / Rpim(I) all: 0.109 / Rrim(I) all: 0.194 / Rsym value: 0.16 / Net I/av σ(I): 4.824 / Net I/σ(I): 10.1 / Num. measured all: 685808 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 反射 シェル | Diffraction-ID: 1 / Rejects: _
|
-位相決定
| 位相決定 | 手法:  分子置換 分子置換 | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| Phasing MR | Model details: Phaser MODE: MR_AUTO
|
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 3B07 prepared using CHAINSAW 解像度: 2.8→48 Å / Cor.coef. Fo:Fc: 0.906 / Cor.coef. Fo:Fc free: 0.856 / WRfactor Rfree: 0.2318 / WRfactor Rwork: 0.1798 / FOM work R set: 0.6812 / SU B: 20.998 / SU ML: 0.362 / SU R Cruickshank DPI: 0.512 / SU Rfree: 0.3468 / 交差検証法: THROUGHOUT / σ(F): 0 / ESU R: 0.512 / ESU R Free: 0.347 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: U VALUES : REFINED INDIVIDUALLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso max: 132.12 Å2 / Biso mean: 36.001 Å2 / Biso min: 2 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: final / 解像度: 2.8→48 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.8→2.869 Å
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj
