+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4qmo | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | MST3 IN COMPLEX WITH Imidazolo-oxindole PKR inhibitor C16 | ||||||
 要素 要素 | Serine/threonine-protein kinase 24 | ||||||
 キーワード キーワード | Transferase/transferase inhibitor / PROTEIN KINASE / MST3 / STK24 / STERILE 20-LIKE KINASE / ATP-BINDING / NUCLEOTIDE-BINDING / PHOSPHOPROTEIN / SERINE/THREONINE-TRANSFERASE / Transferase-transferase inhibitor complex | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報Apoptotic execution phase / FAR/SIN/STRIPAK complex / regulation of axon regeneration / intrinsic apoptotic signaling pathway in response to oxidative stress / execution phase of apoptosis / Apoptotic cleavage of cellular proteins / negative regulation of cell migration / cellular response to starvation / protein autophosphorylation / cellular response to oxidative stress ...Apoptotic execution phase / FAR/SIN/STRIPAK complex / regulation of axon regeneration / intrinsic apoptotic signaling pathway in response to oxidative stress / execution phase of apoptosis / Apoptotic cleavage of cellular proteins / negative regulation of cell migration / cellular response to starvation / protein autophosphorylation / cellular response to oxidative stress / protein phosphorylation / non-specific serine/threonine protein kinase / protein kinase activity / intracellular signal transduction / cadherin binding / protein serine kinase activity / protein serine/threonine kinase activity / nucleolus / Golgi apparatus / signal transduction / extracellular exosome / nucleoplasm / ATP binding / metal ion binding / nucleus / membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  分子置換 / 解像度: 1.898 Å 分子置換 / 解像度: 1.898 Å | ||||||
 データ登録者 データ登録者 | Olesen, S.H. / Watts, C. / Zhu, J.-Y. / Schonbrunn, E. | ||||||
 引用 引用 |  ジャーナル: Chemmedchem / 年: 2016 ジャーナル: Chemmedchem / 年: 2016タイトル: Discovery of Diverse Small-Molecule Inhibitors of Mammalian Sterile20-like Kinase 3 (MST3). 著者: Olesen, S.H. / Zhu, J.Y. / Martin, M.P. / Schonbrunn, E. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4qmo.cif.gz 4qmo.cif.gz | 80.6 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4qmo.ent.gz pdb4qmo.ent.gz | 58.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4qmo.json.gz 4qmo.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4qmo_validation.pdf.gz 4qmo_validation.pdf.gz | 750.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4qmo_full_validation.pdf.gz 4qmo_full_validation.pdf.gz | 753.2 KB | 表示 | |
| XML形式データ |  4qmo_validation.xml.gz 4qmo_validation.xml.gz | 15.8 KB | 表示 | |
| CIF形式データ |  4qmo_validation.cif.gz 4qmo_validation.cif.gz | 22.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qm/4qmo https://data.pdbj.org/pub/pdb/validation_reports/qm/4qmo ftp://data.pdbj.org/pub/pdb/validation_reports/qm/4qmo ftp://data.pdbj.org/pub/pdb/validation_reports/qm/4qmo | HTTPS FTP |
-関連構造データ
| 関連構造データ |  4qmlC  4qmmC  4qmnC  4qmpC  4qmqC  4qmsC  4qmtC  4qmuC  4qmvC  4qmwC  4qmxC  4qmyC  4qmzC  4qnaC  4qo9C  3ckwS  4qmr C: 同じ文献を引用 ( S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | monomer per ASU |
- 要素
要素
-タンパク質 , 1種, 1分子 A
| #1: タンパク質 | 分子量: 35023.934 Da / 分子数: 1 / 断片: RESIDUES 1-303 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: MST3, STK24, STK3 / プラスミド: PET28A / 発現宿主: Homo sapiens (ヒト) / 遺伝子: MST3, STK24, STK3 / プラスミド: PET28A / 発現宿主:  参照: UniProt: Q9Y6E0, non-specific serine/threonine protein kinase |
|---|
-非ポリマー , 6種, 253分子 
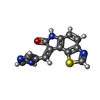









| #2: 化合物 | ChemComp-EDO / |
|---|---|
| #3: 化合物 | ChemComp-34L / ( |
| #4: 化合物 | ChemComp-PG4 / |
| #5: 化合物 | ChemComp-DMS / |
| #6: 化合物 | ChemComp-CL / |
| #7: 水 | ChemComp-HOH / |
-詳細
| Has protein modification | Y |
|---|---|
| 配列の詳細 | THE CRYSTALLIZ |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.56 Å3/Da / 溶媒含有率: 52.01 % |
|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 7.5 詳細: 12.5 mg/mL MST3, 1 mM C16, 25 mM TRIS, PH 8.0, 50 MM HEPES pH 7.5, 125 mM SODIUM CHLORIDE, 100 mM MAGNESIUM CHLORIDE, 15% PEG 400, VAPOR DIFFUSION, HANGING DROP, temperature 291K |
-データ収集
| 回折 | 平均測定温度: 93 K | |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54178 / 波長: 1.54 Å 回転陽極 / タイプ: RIGAKU MICROMAX-007 HF / 波長: 1.54178 / 波長: 1.54 Å | |||||||||
| 検出器 | タイプ: RIGAKU SATURN 944+ / 検出器: CCD / 日付: 2012年8月30日 / 詳細: MIRRORS | |||||||||
| 放射 | モノクロメーター: MIRRORS / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | |||||||||
| 放射波長 |
| |||||||||
| 反射 | 解像度: 1.9→20 Å / Num. obs: 27646 / % possible obs: 98.4 % / Observed criterion σ(I): -3 / 冗長度: 3.5 % / Rsym value: 0.044 / Net I/σ(I): 37.7 | |||||||||
| 反射 シェル | 解像度: 1.9→1.93 Å / 冗長度: 3.3 % / Mean I/σ(I) obs: 5.5 / Rsym value: 0.388 / % possible all: 96 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: 3CKW 解像度: 1.898→19.897 Å / SU ML: 0.2 / σ(F): 1.34 / 位相誤差: 21.57 / 立体化学のターゲット値: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.898→19.897 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj





