| 登録情報 | データベース: PDB / ID: 4o6x
|
|---|
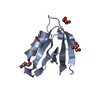
| タイトル | Crystal structure of human Ankyrin G death domain |
|---|
 要素 要素 | Ankyrin-3 |
|---|
 キーワード キーワード | PROTEIN BINDING / alpha helix bundle / death domain / alpha helix |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of cell communication by electrical coupling / positive regulation of membrane depolarization during cardiac muscle cell action potential / maintenance of protein location in plasma membrane / positive regulation of sodium ion transmembrane transporter activity / membrane assembly / protein localization to axon / spectrin-associated cytoskeleton / regulation of potassium ion transport / magnesium ion homeostasis / positive regulation of membrane potential ...positive regulation of cell communication by electrical coupling / positive regulation of membrane depolarization during cardiac muscle cell action potential / maintenance of protein location in plasma membrane / positive regulation of sodium ion transmembrane transporter activity / membrane assembly / protein localization to axon / spectrin-associated cytoskeleton / regulation of potassium ion transport / magnesium ion homeostasis / positive regulation of membrane potential / plasma membrane organization / negative regulation of delayed rectifier potassium channel activity / positive regulation of cation channel activity / cytoskeletal anchor activity / positive regulation of homotypic cell-cell adhesion / positive regulation of sodium ion transport / axon initial segment / costamere / cellular response to magnesium ion / Golgi to plasma membrane protein transport / node of Ranvier / Interaction between L1 and Ankyrins / spectrin binding / neuromuscular junction development / mitotic cytokinesis / lateral plasma membrane / intercalated disc / positive regulation of protein targeting to membrane / neuronal action potential / cytoskeletal protein binding / COPI-mediated anterograde transport / T-tubule / axonogenesis / basal plasma membrane / sarcoplasmic reticulum / protein localization to plasma membrane / neuromuscular junction / establishment of protein localization / sarcolemma / structural constituent of cytoskeleton / Z disc / protein-macromolecule adaptor activity / basolateral plasma membrane / transmembrane transporter binding / postsynaptic membrane / neuron projection / lysosome / cadherin binding / dendrite / positive regulation of gene expression / cell surface / endoplasmic reticulum / Golgi apparatus / signal transduction / plasma membrane / cytosol類似検索 - 分子機能 Ankyrin-3, death domain / Ankyrin, UPA domain / UPA domain / : / Domain present in ZO-1 and Unc5-like netrin receptors / ZU5 domain / ZU5 domain / ZU5 domain profile. / Death Domain, Fas / Death Domain, Fas ...Ankyrin-3, death domain / Ankyrin, UPA domain / UPA domain / : / Domain present in ZO-1 and Unc5-like netrin receptors / ZU5 domain / ZU5 domain / ZU5 domain profile. / Death Domain, Fas / Death Domain, Fas / Ankyrin repeats (many copies) / Death domain profile. / DEATH domain, found in proteins involved in cell death (apoptosis). / Death domain / Death domain / Death-like domain superfamily / Ankyrin repeat / Ankyrin repeats (3 copies) / Ankyrin repeat profile. / Ankyrin repeat region circular profile. / ankyrin repeats / Ankyrin repeat / Ankyrin repeat-containing domain superfamily / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.103 Å 分子置換 / 解像度: 2.103 Å |
|---|
 データ登録者 データ登録者 | Liu, Y. / Zhang, Y. / Wang, J.H. |
|---|
 引用 引用 |  ジャーナル: Proteins / 年: 2014 ジャーナル: Proteins / 年: 2014
タイトル: Crystal structure of human Ankyrin G death domain.
著者: Liu, Y. / Zhang, Y. / Wang, J.H. |
|---|
| 履歴 | | 登録 | 2013年12月24日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2014年10月29日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2014年12月17日 | Group: Database references |
|---|
| 改定 1.2 | 2015年2月11日 | Group: Other |
|---|
| 改定 1.3 | 2023年9月20日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.103 Å
分子置換 / 解像度: 2.103 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proteins / 年: 2014
ジャーナル: Proteins / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4o6x.cif.gz
4o6x.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4o6x.ent.gz
pdb4o6x.ent.gz PDB形式
PDB形式 4o6x.json.gz
4o6x.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4o6x_validation.pdf.gz
4o6x_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4o6x_full_validation.pdf.gz
4o6x_full_validation.pdf.gz 4o6x_validation.xml.gz
4o6x_validation.xml.gz 4o6x_validation.cif.gz
4o6x_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/o6/4o6x
https://data.pdbj.org/pub/pdb/validation_reports/o6/4o6x ftp://data.pdbj.org/pub/pdb/validation_reports/o6/4o6x
ftp://data.pdbj.org/pub/pdb/validation_reports/o6/4o6x
 リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: ANK3, Ankyrin G / プラスミド: pET21b / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: ANK3, Ankyrin G / プラスミド: pET21b / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL17U / 波長: 0.98 Å
/ ビームライン: BL17U / 波長: 0.98 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj











