| 登録情報 | データベース: PDB / ID: 4nnm
|
|---|
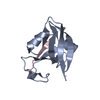
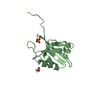
| タイトル | Tax-Interacting Protein-1 (TIP-1) PDZ domain bound to Y-iCAL36 (YPTSII) peptide |
|---|
 要素 要素 | - TIP-1 PDZ domain
- Tax1-binding protein 3
|
|---|
 キーワード キーワード | PROTEIN BINDING / Tax-Interacting Protein-1 / TIP-1 / PDZ / PDZ-peptide |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
negative regulation of protein localization to cell surface / RHO GTPases Activate Rhotekin and Rhophilins / activation of GTPase activity / negative regulation of Wnt signaling pathway / Rho protein signal transduction / beta-catenin binding / Wnt signaling pathway / fibrillar center / actin cytoskeleton / negative regulation of cell population proliferation ...negative regulation of protein localization to cell surface / RHO GTPases Activate Rhotekin and Rhophilins / activation of GTPase activity / negative regulation of Wnt signaling pathway / Rho protein signal transduction / beta-catenin binding / Wnt signaling pathway / fibrillar center / actin cytoskeleton / negative regulation of cell population proliferation / intracellular membrane-bounded organelle / extracellular exosome / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 Tax1-binding protein 3 / : / PDZ domain / Pdz3 Domain / PDZ domain / PDZ domain profile. / Domain present in PSD-95, Dlg, and ZO-1/2. / PDZ domain / PDZ superfamily / Roll / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.6 Å 分子置換 / 解像度: 1.6 Å |
|---|
 データ登録者 データ登録者 | Amacher, J.F. / Madden, D.R. |
|---|
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Published
タイトル: A CAL Inhibitor with Single-PDZ Specificity Rescues deltaF508-CFTR
著者: Cushing, P.R. / Amacher, J.F. / Vouilleme, L. / Culatti, S. / Deng, B. / Gerber, S.A. / Boisguerin, P. / Madden, D.R. |
|---|
| 履歴 | | 登録 | 2013年11月18日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2015年1月21日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2023年9月20日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.6 Å
分子置換 / 解像度: 1.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4nnm.cif.gz
4nnm.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4nnm.ent.gz
pdb4nnm.ent.gz PDB形式
PDB形式 4nnm.json.gz
4nnm.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/nn/4nnm
https://data.pdbj.org/pub/pdb/validation_reports/nn/4nnm ftp://data.pdbj.org/pub/pdb/validation_reports/nn/4nnm
ftp://data.pdbj.org/pub/pdb/validation_reports/nn/4nnm リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 遺伝子: TAX1BP3, TIP1 / プラスミド: pET16b / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: TAX1BP3, TIP1 / プラスミド: pET16b / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X6A / 波長: 0.8856 Å
/ ビームライン: X6A / 波長: 0.8856 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj


