[English] 日本語
 Yorodumi
Yorodumi- PDB-4n6c: Crystal Structure of the B1RZQ2 protein from Streptococcus pneumo... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4n6c | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal Structure of the B1RZQ2 protein from Streptococcus pneumoniae. Northeast Structural Genomics Consortium (NESG) Target SpR36. | ||||||
 Components Components | uncharacterized protein | ||||||
 Keywords Keywords | STRUCTURAL GENOMICS / UNKNOWN FUNCTION / PSI-Biology / Protein Structure Initiative / Northeast Structural Genomics Consortium / NESG / SpR36 / PF08020 / DUF1706 | ||||||
| Function / homology | dinb family like domain / Four Helix Bundle (Hemerythrin (Met), subunit A) / Up-down Bundle / Mainly Alpha / BROMIDE ION / :  Function and homology information Function and homology information | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  SAD / Resolution: 1.548 Å SAD / Resolution: 1.548 Å | ||||||
 Authors Authors | Vorobiev, S. / Seetharaman, J. / Patel, D. / Xiao, R. / Ciccosanti, C. / Wang, D. / Everett, J.K. / Acton, T.B. / Montelione, G.T. / Tong, L. ...Vorobiev, S. / Seetharaman, J. / Patel, D. / Xiao, R. / Ciccosanti, C. / Wang, D. / Everett, J.K. / Acton, T.B. / Montelione, G.T. / Tong, L. / Hunt, J.F. / Northeast Structural Genomics Consortium (NESG) | ||||||
 Citation Citation |  Journal: To be Published Journal: To be PublishedTitle: Crystal Structure of the B1RZQ2 protein from Streptococcus pneumoniae. Authors: Vorobiev, S. / Seetharaman, J. / Patel, D. / Xiao, R. / Ciccosanti, C. / Wang, D. / Everett, J.K. / Acton, T.B. / Montelione, G.T. / Tong, L. / Hunt, J.F. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4n6c.cif.gz 4n6c.cif.gz | 170.5 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4n6c.ent.gz pdb4n6c.ent.gz | 137.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4n6c.json.gz 4n6c.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/n6/4n6c https://data.pdbj.org/pub/pdb/validation_reports/n6/4n6c ftp://data.pdbj.org/pub/pdb/validation_reports/n6/4n6c ftp://data.pdbj.org/pub/pdb/validation_reports/n6/4n6c | HTTPS FTP |
|---|
-Related structure data
| Similar structure data | |
|---|---|
| Other databases |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 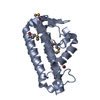
| ||||||||
| 2 | 
| ||||||||
| Unit cell |
| ||||||||
| Details | monomer,23.81 kD,92.1% |
- Components
Components
| #1: Protein | Mass: 22125.416 Da / Num. of mol.: 2 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   #2: Chemical | #3: Water | ChemComp-HOH / | Has protein modification | Y | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.08 Å3/Da / Density % sol: 40.77 % |
|---|---|
| Crystal grow | Temperature: 291 K / Method: microbatch crystallization under oil / pH: 10 Details: 40% PEG 8000, 0.1 M sodium bromide, 0.1 M CAPS, pH 10.0, Microbatch crystallization under oil, temperature 291K |
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  NSLS NSLS  / Beamline: X4C / Wavelength: 0.97907 Å / Beamline: X4C / Wavelength: 0.97907 Å |
| Detector | Type: MAR CCD 165 mm / Detector: CCD / Date: Oct 3, 2013 |
| Radiation | Monochromator: Si 111 CHANNEL / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 0.97907 Å / Relative weight: 1 |
| Reflection | Resolution: 1.548→50 Å / Num. all: 103638 / Num. obs: 100218 / % possible obs: 96.7 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 / Redundancy: 3.7 % / Biso Wilson estimate: 19.13 Å2 / Rmerge(I) obs: 0.081 / Net I/σ(I): 24.4 |
| Reflection shell | Resolution: 1.55→1.61 Å / Redundancy: 3.6 % / Rmerge(I) obs: 0.688 / Mean I/σ(I) obs: 2.1 / Num. unique all: 10387 / % possible all: 94.2 |
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  SAD / Resolution: 1.548→36.946 Å / Occupancy max: 1 / Occupancy min: 0.5 / SU ML: 0.48 / Cross valid method: THROUGHOUT / σ(F): 1.13 / Phase error: 21.43 / Stereochemistry target values: ML SAD / Resolution: 1.548→36.946 Å / Occupancy max: 1 / Occupancy min: 0.5 / SU ML: 0.48 / Cross valid method: THROUGHOUT / σ(F): 1.13 / Phase error: 21.43 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.86 Å / VDW probe radii: 1.1 Å / Solvent model: FLAT BULK SOLVENT MODEL / Bsol: 50.748 Å2 / ksol: 0.41 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 75.7 Å2 / Biso mean: 26.629 Å2 / Biso min: 7.52 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.548→36.946 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 30
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller











 PDBj
PDBj




