| ソフトウェア | | 名称 | バージョン | 分類 | NB |
|---|
| DENZO | | データ削減 | | | SCALEPACK | | データスケーリング | | | SOLVE | | 位相決定 | | | RESOLVE | 2.15 | 位相決定 | | | PHENIX | 1.8.1_1168| 精密化 | | | PDB_EXTRACT | 3.11 | データ抽出 | | | HKL-2000 | | データ収集 | | | HKL-2000 | | データ削減 | | | HKL-2000 | | データスケーリング | | |
|
|---|
| 精密化 | 構造決定の手法:  単波長異常分散 / 解像度: 2.673→20.14 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.7754 / SU ML: 0.39 / σ(F): 1.35 / 位相誤差: 28.44 / 立体化学のターゲット値: ML 単波長異常分散 / 解像度: 2.673→20.14 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.7754 / SU ML: 0.39 / σ(F): 1.35 / 位相誤差: 28.44 / 立体化学のターゲット値: ML
| Rfactor | 反射数 | %反射 |
|---|
| Rfree | 0.2427 | 1151 | 10.02 % |
|---|
| Rwork | 0.201 | - | - |
|---|
| obs | 0.2051 | 11484 | 95.71 % |
|---|
|
|---|
| 溶媒の処理 | 減衰半径: 0.9 Å / VDWプローブ半径: 1.11 Å / 溶媒モデル: FLAT BULK SOLVENT MODEL |
|---|
| 原子変位パラメータ | Biso max: 205.22 Å2 / Biso mean: 81.9439 Å2 / Biso min: 33.85 Å2 |
|---|
| 精密化ステップ | サイクル: LAST / 解像度: 2.673→20.14 Å
| タンパク質 | 核酸 | リガンド | 溶媒 | 全体 |
|---|
| 原子数 | 0 | 2472 | 2 | 25 | 2499 |
|---|
|
|---|
| 拘束条件 | | Refine-ID | タイプ | Dev ideal | 数 |
|---|
| X-RAY DIFFRACTION | f_bond_d| 0.001 | 2762 | | X-RAY DIFFRACTION | f_angle_d| 0.409 | 4308 | | X-RAY DIFFRACTION | f_chiral_restr| 0.019 | 568 | | X-RAY DIFFRACTION | f_plane_restr| 0.002 | 114 | | X-RAY DIFFRACTION | f_dihedral_angle_d| 11.385 | 1366 | | | | | |
|
|---|
| LS精密化 シェル | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 8 | 解像度 (Å) | Rfactor Rfree | Num. reflection Rfree | Rfactor Rwork | Num. reflection Rwork | Num. reflection all | % reflection obs (%) |
|---|
| 2.6732-2.7945 | 0.3642 | 118 | 0.3358 | 1054 | 1172 | 79 | | 2.7945-2.9414 | 0.3948 | 139 | 0.322 | 1257 | 1396 | 93 | | 2.9414-3.1251 | 0.3302 | 145 | 0.2628 | 1304 | 1449 | 99 | | 3.1251-3.3654 | 0.2522 | 144 | 0.2216 | 1304 | 1448 | 98 | | 3.3654-3.7022 | 0.2475 | 148 | 0.1883 | 1310 | 1458 | 97 | | 3.7022-4.2335 | 0.2074 | 151 | 0.1636 | 1347 | 1498 | 100 | | 4.2335-5.3175 | 0.2261 | 151 | 0.1738 | 1364 | 1515 | 100 | | 5.3175-20.1403 | 0.1969 | 155 | 0.1767 | 1393 | 1548 | 99 |
|
|---|
| 精密化 TLS | 手法: refined / Refine-ID: X-RAY DIFFRACTION | ID | L11 (°2) | L12 (°2) | L13 (°2) | L22 (°2) | L23 (°2) | L33 (°2) | S11 (Å °) | S12 (Å °) | S13 (Å °) | S21 (Å °) | S22 (Å °) | S23 (Å °) | S31 (Å °) | S32 (Å °) | S33 (Å °) | T11 (Å2) | T12 (Å2) | T13 (Å2) | T22 (Å2) | T23 (Å2) | T33 (Å2) | Origin x (Å) | Origin y (Å) | Origin z (Å) |
|---|
| 1 | 1.1106 | 1.052 | -0.1352 | 0.8831 | -0.7675 | 1.5182 | -0.7386 | 1.4023 | 0.3415 | -0.528 | -0.0318 | 0.1217 | -0.6032 | -0.4585 | 0.5808 | 1.2493 | 0.0238 | -0.3625 | 1.4765 | -0.1706 | 0.6877 | 16.661 | 49.9943 | -8.718 | | 2 | 3.6283 | 0.2261 | 1.172 | 5.3467 | -1.2178 | 0.9388 | -0.1027 | 0.2973 | 0.1901 | 0.1058 | 0.0199 | 0.1957 | -0.1163 | 0.0397 | 0.0959 | 0.6234 | 0.0244 | -0.1886 | 0.3289 | -0.0663 | 0.4647 | 14.6338 | 45.6602 | 12.1494 | | 3 | 4.8434 | 1.5956 | -2.3259 | 4.7993 | 2.1744 | 4.5734 | -0.8383 | 0.9265 | 0.8614 | -0.6443 | 1.242 | 0.3721 | 0.095 | -0.7815 | 0.2788 | 1.4041 | 0.0416 | -0.5583 | 1.6596 | -0.0917 | 0.7231 | 10.8361 | 50.0262 | -21.7703 | | 4 | 3.0535 | -0.0068 | 0.9871 | 2.9871 | -0.5061 | 0.389 | -0.6597 | -0.0276 | -0.1546 | -0.1952 | 0.5633 | 0.0058 | -0.301 | 1.5212 | 0.0341 | 1.4957 | -0.3037 | 0.0132 | 2.0283 | -0.0993 | 0.549 | 36.7448 | 15.2994 | 50.0423 | | 5 | 1.7832 | 0.9028 | -1.7381 | 2.6998 | -2.9547 | 3.5887 | 0.3214 | -0.7736 | -0.1093 | 0.6981 | -0.1749 | -0.0449 | -0.7013 | 0.7871 | 0.003 | 0.9211 | -0.0804 | -0.0975 | 0.6205 | -0.0009 | 0.4858 | 28.4895 | 19.4721 | 29.9598 | | 6 | 2.2348 | -1.7207 | 0.1968 | 5.804 | -2.4489 | 2.5524 | -0.0504 | -0.2493 | -0.1786 | 0.5593 | 0.2031 | 0.2963 | 0.0353 | -0.3017 | -0.1244 | 0.7165 | -0.0672 | -0.1304 | 0.3402 | -0.0057 | 0.3874 | 16.5223 | 23.2008 | 23.2747 | | 7 | 2.1831 | -0.4185 | 2.3671 | 2.5398 | -0.261 | 2.6442 | -0.0412 | -1.1809 | 0.0334 | 0.9528 | 0.3591 | 0.032 | -0.4755 | 1.0078 | -0.0006 | 1.4719 | -0.3737 | -0.2032 | 1.6357 | -0.0498 | 0.4283 | 34.2646 | 15.3918 | 47.799 |
|
|---|
| 精密化 TLSグループ | | ID | Refine-ID | Refine TLS-ID | Selection details | Auth asym-ID | Auth seq-ID |
|---|
| 1 | X-RAY DIFFRACTION | 1 | chain 'A' and (resid 24 through 40 )A| 0 | | 2 | X-RAY DIFFRACTION | 2 | chain 'A' and (resid 41 through 73 )A| 0 | | 3 | X-RAY DIFFRACTION | 3 | chain 'A' and (resid 74 through 80 )A| 0 | | 4 | X-RAY DIFFRACTION | 4 | chain 'B' and (resid 24 through 32 )B| 0 | | 5 | X-RAY DIFFRACTION | 5 | chain 'B' and (resid 33 through 40 )B| 0 | | 6 | X-RAY DIFFRACTION | 6 | chain 'B' and (resid 41 through 68 )B| 0 | | 7 | X-RAY DIFFRACTION | 7 | chain 'B' and (resid 69 through 80 )B| 0 | | | | | | | | | | | | | | |
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 2.673 Å
単波長異常分散 / 解像度: 2.673 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2013
ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4jrc.cif.gz
4jrc.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4jrc.ent.gz
pdb4jrc.ent.gz PDB形式
PDB形式 4jrc.json.gz
4jrc.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4jrc_validation.pdf.gz
4jrc_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4jrc_full_validation.pdf.gz
4jrc_full_validation.pdf.gz 4jrc_validation.xml.gz
4jrc_validation.xml.gz 4jrc_validation.cif.gz
4jrc_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/jr/4jrc
https://data.pdbj.org/pub/pdb/validation_reports/jr/4jrc ftp://data.pdbj.org/pub/pdb/validation_reports/jr/4jrc
ftp://data.pdbj.org/pub/pdb/validation_reports/jr/4jrc リンク
リンク 集合体
集合体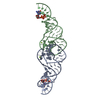


 要素
要素 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  CHESS
CHESS  / ビームライン: A1 / 波長: 0.9767 Å
/ ビームライン: A1 / 波長: 0.9767 Å 単波長異常分散
単波長異常分散 解析
解析 単波長異常分散 / 解像度: 2.673→20.14 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.7754 / SU ML: 0.39 / σ(F): 1.35 / 位相誤差: 28.44 / 立体化学のターゲット値: ML
単波長異常分散 / 解像度: 2.673→20.14 Å / Occupancy max: 1 / Occupancy min: 1 / FOM work R set: 0.7754 / SU ML: 0.39 / σ(F): 1.35 / 位相誤差: 28.44 / 立体化学のターゲット値: ML ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
































