ソフトウェア 名称 バージョン 分類 NB 0.1.27データスケーリング Show 位相決定 Show dev_1148精密化 Show 3.11 データ抽出 Show データ収集 データ削減
精密化 構造決定の手法 開始モデル 解像度 / Occupancy max / Occupancy min / SU ML / σ(F) / 位相誤差 / 立体化学のターゲット値 Rfactor 反射数 %反射 Selection details Rfree 0.1901 2761 5.09 % RANDOM Rwork 0.1611 - - - obs 0.1626 54268 99.99 % -
溶媒の処理 減衰半径 / VDWプローブ半径 / 溶媒モデル 原子変位パラメータ Biso max 2 / Biso mean 2 / Biso min 2 精密化ステップ サイクル / 解像度 タンパク質 核酸 リガンド 溶媒 全体 原子数 2703 0 77 259 3039
拘束条件 Refine-ID タイプ Dev ideal 数 X-RAY DIFFRACTION f_bond_d0.009 2897 X-RAY DIFFRACTION f_angle_d1.046 3895 X-RAY DIFFRACTION f_chiral_restr0.083 405 X-RAY DIFFRACTION f_plane_restr0.008 514 X-RAY DIFFRACTION f_dihedral_angle_d14.926 1092
LS精密化 シェル Refine-ID / Total num. of bins used / % reflection obs
大きな表を表示 (6 x 20) 大きな表を隠す 解像度 (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Num. reflection all 1.6-1.6276 0.2552 141 0.2386 2554 2695 1.6276-1.6572 0.2772 128 0.2271 2583 2711 1.6572-1.6891 0.2443 150 0.2295 2521 2671 1.6891-1.7235 0.2367 119 0.2168 2611 2730 1.7235-1.761 0.237 149 0.2002 2550 2699 1.761-1.802 0.2176 161 0.2001 2553 2714 1.802-1.8471 0.2 140 0.1771 2567 2707 1.8471-1.897 0.2109 144 0.164 2549 2693 1.897-1.9528 0.1893 141 0.1638 2571 2712 1.9528-2.0158 0.2143 142 0.1608 2569 2711 2.0158-2.0879 0.179 134 0.1534 2593 2727 2.0879-2.1715 0.1788 132 0.1484 2561 2693 2.1715-2.2703 0.1714 145 0.142 2588 2733 2.2703-2.39 0.153 140 0.1453 2563 2703 2.39-2.5397 0.1789 123 0.1529 2593 2716 2.5397-2.7357 0.1874 143 0.1587 2567 2710 2.7357-3.0109 0.1923 144 0.1713 2590 2734 3.0109-3.4463 0.1974 130 0.1649 2579 2709 3.4463-4.341 0.155 124 0.1409 2611 2735 4.341-37.5458 0.2054 131 0.1581 2634 2765
精密化 TLS 手法 / Refine-ID
大きな表を表示 (25 x 13) 大きな表を隠す ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 2.0926 0.7549 1.2064 1.4506 0.1966 2.2461 -0.0313 0.3221 -0.1137 -0.2019 0.0305 -0.0546 0.13 0.091 -0.0206 0.1732 0.0049 0.0239 0.1752 -0.0176 0.1756 63.5456 -18.0081 -5.6587 2 2.2122 -0.5059 0.7233 2.0612 -0.0572 1.6372 -0.0296 0.0894 -0.0944 -0.0614 -0.0023 0.3077 0.0344 -0.1702 0.0194 0.1258 -0.0313 -0.0051 0.1544 -0.0066 0.1971 51.3688 -15.5573 -0.8437 3 6.2632 -3.6784 -0.6633 4.3467 0.6797 3.2035 0.0286 0.5162 0.4758 -0.4543 -0.0157 0.0414 -0.2344 0.0042 0.0085 0.2152 -0.0484 -0.0333 0.1922 0.0438 0.2572 53.1268 -5.3934 -8.6934 4 2.1876 1.0991 1.569 4.3008 5.4425 6.9361 -0.0072 0.1245 -0.0062 -0.2709 0.0271 -0.1209 -0.1011 -0.0568 -0.0697 0.159 -0.0164 0.0034 0.1505 0.0119 0.1684 63.8716 -6.0932 -5.184 5 4.7738 0.7624 -0.5159 2.3271 -0.6796 3.7464 0.0107 -0.1139 -0.0893 0.0702 -0.1427 -0.2697 0.0413 0.2972 0.132 0.1147 -0.011 -0.0138 0.1455 -0.0093 0.1532 71.9764 -12.6038 6.4048 6 0.8122 0.233 -0.3204 1.591 -1.0615 2.3945 -0.1438 -0.5346 -0.1685 0.4507 0.1134 0.2404 0.0626 0.0481 -0.1512 0.2596 0.0532 0.0829 0.2746 0.068 0.17 61.1551 -15.0337 31.4844 7 3.5043 0.9711 0.0185 0.3479 -0.4557 2.7004 0.1175 -0.2538 -0.4083 0.5206 0.3227 0.6532 -0.0612 -0.1985 -0.0047 0.3169 0.0114 0.0324 0.184 0.1327 0.3498 67.6338 -35.6799 28.1723 8 0.9942 -0.3281 0.7842 0.7742 -0.584 1.407 0.047 -0.2828 -0.1968 0.2694 0.1246 0.265 0.1125 -0.0685 -0.1275 0.2457 0.0039 0.0412 0.2112 0.0874 0.2106 67.7914 -28.9096 31.787 9 1.9326 -0.1536 0.7879 1.8162 -0.4294 1.4612 0.1096 -0.0935 -0.1434 0.0813 0.0517 0.3084 0.0722 -0.1 -0.1562 0.2064 -0.0087 0.0142 0.1928 0.0608 0.2117 61.5265 -25.0188 23.8324 10 3.4222 -1.5706 2.0722 1.6242 -0.4284 1.5564 -0.0293 -0.0696 -0.0051 0.2829 -0.5157 -0.6493 -0.0422 1.0659 0.3842 0.2168 -0.0006 -0.0717 0.4812 0.206 0.297 81.4492 -25.2901 27.1467 11 2.2436 -1.1113 1.1975 3.451 0.7116 3.6646 -0.009 -0.8341 -0.4873 0.7855 -0.0931 -0.2444 -0.5665 0.3935 0.1411 0.4072 -0.0898 -0.0413 0.3575 0.1152 0.2031 76.2454 -22.924 35.1025 12 8.9367 2.5634 -7.1396 5.4768 -5.22 8.4634 -0.1483 -0.4518 0.2228 0.5649 0.0221 -0.1515 -0.291 0.0115 0.134 0.2681 -0.0157 -0.0491 0.2287 -0.0046 0.1208 72.719 -12.7656 31.7147 13 1.6711 0.026 0.5673 5.3027 0.3097 4.1583 -0.0787 -0.1218 0.3201 0.0961 -0.0831 0.2787 -0.2249 -0.0944 0.1812 0.1772 0.0041 0.0227 0.1177 0.0195 0.1324 64.1308 -6.914 20.1714
精密化 TLSグループ ID Refine-ID Refine TLS-ID Selection details Auth asym-ID Auth seq-ID 1 X-RAY DIFFRACTION 1 CHAIN 'A' AND (RESID 2 THROUGH 41 )A0 2 X-RAY DIFFRACTION 2 CHAIN 'A' AND (RESID 42 THROUGH 130 )A0 3 X-RAY DIFFRACTION 3 CHAIN 'A' AND (RESID 131 THROUGH 148 )A0 4 X-RAY DIFFRACTION 4 CHAIN 'A' AND (RESID 149 THROUGH 160 )A0 5 X-RAY DIFFRACTION 5 CHAIN 'A' AND (RESID 161 THROUGH 180 )A0 6 X-RAY DIFFRACTION 6 CHAIN 'B' AND (RESID 2 THROUGH 37 )B0 7 X-RAY DIFFRACTION 7 CHAIN 'B' AND (RESID 38 THROUGH 53 )B0 8 X-RAY DIFFRACTION 8 CHAIN 'B' AND (RESID 54 THROUGH 82 )B0 9 X-RAY DIFFRACTION 9 CHAIN 'B' AND (RESID 83 THROUGH 117 )B0 10 X-RAY DIFFRACTION 10 CHAIN 'B' AND (RESID 118 THROUGH 130 )B0 11 X-RAY DIFFRACTION 11 CHAIN 'B' AND (RESID 131 THROUGH 148 )B0 12 X-RAY DIFFRACTION 12 CHAIN 'B' AND (RESID 149 THROUGH 160 )B0 13 X-RAY DIFFRACTION 13 CHAIN 'B' AND (RESID 161 THROUGH 180 )B
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.6 Å
分子置換 / 解像度: 1.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 2013
ジャーナル: Biochemistry / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4jes.cif.gz
4jes.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4jes.ent.gz
pdb4jes.ent.gz PDB形式
PDB形式 4jes.json.gz
4jes.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/je/4jes
https://data.pdbj.org/pub/pdb/validation_reports/je/4jes ftp://data.pdbj.org/pub/pdb/validation_reports/je/4jes
ftp://data.pdbj.org/pub/pdb/validation_reports/je/4jes リンク
リンク 集合体
集合体
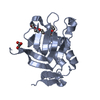

 要素
要素




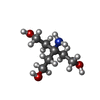





 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 17-ID / 波長: 1 Å
/ ビームライン: 17-ID / 波長: 1 Å 分子置換
分子置換 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


















 PDBj
PDBj


