Software Name Version Classification data collectionphasing(phenix.refine: 1.7.3_928)refinementdata reductiondata scaling
Refinement Method to determine structure Starting model Resolution / SU ML / σ(F) / Phase error / Stereochemistry target values Rfactor Num. reflection % reflection Selection details Rfree 0.2744 1206 5.07 % RANDOM Rwork 0.2148 - - - obs 0.2178 23808 91.17 % -
Solvent computation Shrinkage radii / VDW probe radii / Solvent model / Bsol 2 / ksol 3 Displacement parameters Baniso -1 Baniso -2 Baniso -3 1- -2.7892 Å2 0 Å2 -0 Å2 2- - 6.1624 Å2 0 Å2 3- - - -0.8828 Å2
Refinement step Cycle / Resolution Protein Nucleic acid Ligand Solvent Total Num. atoms 3185 0 0 211 3396
Refine LS restraints Refine-ID Type Dev ideal Number X-RAY DIFFRACTION f_bond_d0.01 3362 X-RAY DIFFRACTION f_angle_d1.397 4581 X-RAY DIFFRACTION f_dihedral_angle_d13.992 1187 X-RAY DIFFRACTION f_chiral_restr0.083 487 X-RAY DIFFRACTION f_plane_restr0.005 605
Refine LS restraints NCS Ens-ID Dom-ID Auth asym-ID Number Refine-ID Type Rms dev position (Å)1 1 A1450 X-RAY DIFFRACTION POSITIONAL1 2 B1450 X-RAY DIFFRACTION POSITIONAL0.063
LS refinement shell Resolution (Å)Rfactor Rfree Num. reflection Rfree Rfactor Rwork Num. reflection Rwork Refine-ID % reflection obs (%)2-2.0801 0.3324 126 0.2163 2575 X-RAY DIFFRACTION 95 2.0801-2.1748 0.2812 139 0.1962 2526 X-RAY DIFFRACTION 93 2.1748-2.2894 0.2907 161 0.2111 2410 X-RAY DIFFRACTION 90 2.2894-2.4329 0.2873 126 0.2071 2608 X-RAY DIFFRACTION 95 2.4329-2.6207 0.2932 124 0.2162 2521 X-RAY DIFFRACTION 92 2.6207-2.8843 0.3298 148 0.2092 2458 X-RAY DIFFRACTION 90 2.8843-3.3015 0.2423 127 0.2142 2567 X-RAY DIFFRACTION 93 3.3015-4.1589 0.2266 127 0.1978 2487 X-RAY DIFFRACTION 89 4.1589-40.2684 0.2675 128 0.25 2450 X-RAY DIFFRACTION 84
Refinement TLS params. Method / Refine-ID
Show large table (25 x 40) Hide large table ID L11 (°2 )L12 (°2 )L13 (°2 )L22 (°2 )L23 (°2 )L33 (°2 )S11 (Å °)S12 (Å °)S13 (Å °)S21 (Å °)S22 (Å °)S23 (Å °)S31 (Å °)S32 (Å °)S33 (Å °)T11 (Å2 )T12 (Å2 )T13 (Å2 )T22 (Å2 )T23 (Å2 )T33 (Å2 )Origin x (Å)Origin y (Å)Origin z (Å)1 0.3136 0.2354 0.1907 0.236 0.0445 0.2834 -0.0105 0.0743 -0.0733 -0.0314 0.0268 -0.0743 0.0155 0.0536 -0.0797 0.0368 0.0011 0.0654 0.1156 -0.147 0.1586 -18.7464 -21.3027 -5.9755 2 0.2067 -0.1393 0.2523 0.3701 0.4735 1.8502 -0.1041 -0.127 -0.0182 0.1408 0.0998 0.0227 0.0783 0.0734 -0.1299 0.0502 0.0529 0.0102 0.0503 0.0434 0.0314 -19.0532 -20.9134 17.4881 3 0.8453 -0.3125 -0.4948 1.5786 1.0882 1.7525 -0.0694 -0.0474 -0.0423 0.1311 0.0277 0.0226 0.1736 0.139 0.0267 0.0952 0.0669 0.0758 0.079 0.0644 0.0235 -20.667 -23.5952 22.6596 4 0.0963 0.0436 0.1047 0.0744 -0.0554 0.6485 -0.0149 0.037 -0.0091 -0.0356 -0.0221 0.0191 0.0022 -0.0462 -0.0551 0.0282 -0.0344 -0.0223 0.0673 -0.0636 -0.0527 -23.6408 -18.6119 0.9423 5 2.7606 1.1458 -1.2864 1.3127 -1.0361 1.2831 0.0008 0.1063 0.0911 -0.0576 0.0752 0.0531 0.0358 -0.0653 0.0396 0.0167 -0.018 -0.0441 0.0919 0.0649 0.1283 -24.8866 -14.0108 2.0526 6 0.1018 0.158 -0.0413 0.2639 -0.0853 0.0308 0.0484 -0.0913 -0.0034 0.0773 -0.0018 0.0305 -0.0502 0.017 0.1113 0.0017 -0.041 0.0539 0.0936 0.1004 0.0345 -26.7722 -21.41 21.8555 7 0.1117 0.197 -0.0046 0.4911 0.3162 0.7199 -0.0619 -0.0586 -0.0305 -0.0625 0.0836 -0.0781 -0.0356 0.0358 -0.0033 0.0154 0.0264 -0.0139 0.0499 -0.0066 0.0234 -20.623 -12.9664 14.1491 8 0.5082 0.0065 0.0829 0.2705 -0.0683 0.4258 0.0916 0.0649 -0.0359 -0.0503 0.0387 -0.0092 -0.003 0.0158 0.2198 0.0189 0.0481 0.0241 -0.0978 0.0034 0.1102 -10.2919 -13.4137 -1.9134 9 0.7311 -0.6357 -0.8064 1.8481 1.4329 2.0405 -0.0318 -0.159 0.1672 0.101 0.1093 -0.053 -0.0931 0.0351 -0.0443 0.0448 0.0409 0.0061 0.0724 -0.098 0.1002 -12.1749 -7.7805 19.3062 10 0.3309 -0.331 -0.3377 0.768 0.6224 0.532 -0.0827 -0.1229 0.1021 0.1315 0.11 0.0099 -0.0066 0.0207 0.0322 0.0951 0.069 -0.0259 0.0651 -0.0115 0.0608 -11.6706 -12.2531 20.8211 11 0.3063 -0.4679 -0.2677 1.1048 0.2399 0.3079 0.0541 0.0525 -0.0679 -0.0143 0.0072 0.0455 0.067 0.0075 -0.0458 0.1544 0.0853 -0.0789 0.0417 -0.0458 0.1828 -12.4637 -25.4104 4.6893 12 0.5707 -0.2332 0.0933 0.304 -0.2871 0.3085 -0.046 -0.132 -0.0329 0.0618 0.0304 0.0542 -0.0482 0.0003 -0.113 0.0694 0.0473 0.0247 0.0502 0.0265 0.0798 -15.5047 -19.9568 14.1611 13 1.0037 -0.2752 -0.0378 0.1131 -0.0506 0.1022 -0.0669 -0.1213 0.0819 0.1162 0.0787 -0.0292 -0.0692 -0.0084 0.0891 0.1687 0.1187 -0.0278 0.1194 -0.0269 0.0587 -22.8687 -6.3594 31.6432 14 0.1615 0.0508 -0.0752 0.1105 -0.2173 0.4245 -0.0125 0.0101 0.0075 0.0562 0.0266 0.0261 -0.0642 -0.0691 -0.0855 0.0609 0.1943 -0.0118 0.1313 0.0075 0.0621 -25.9246 -3.4184 17.3209 15 0.3286 -0.0723 -0.1148 0.2491 0.0429 0.1317 -0.0423 -0.0227 0.0546 0.1056 0.018 0.0567 -0.0935 -0.0362 -0.0765 -0.0292 0.1148 0.0279 -0.1042 -0.046 0.0557 -11.1708 -3.1433 10.9425 16 0.0145 0.0566 -0.0408 0.3557 0.0433 0.4425 0.0425 -0.0458 0.0995 0.1104 0.03 0.1462 -0.1245 -0.0974 0.2487 -0.0243 0.1374 0.0881 0.0771 -0.138 0.1009 -17.597 -2.5791 18.2687 17 0.3948 -0.1723 -0.0764 0.7151 -0.0038 0.2289 -0.0205 0.0098 0.017 0.0096 -0.0061 0.0451 -0.079 -0.0806 -0.0095 0.0445 0.0331 0.0084 0.0243 -0.0015 0.0615 -5.0061 -10.2085 1.1776 18 0.669 0.3053 -0.3828 0.6589 -0.1954 0.3746 -0.0628 -0.0105 -0.0488 0.0352 -0.0152 0.0317 0.0551 -0.0345 -0.1424 0.046 0.0535 0.0661 0.0043 -0.0378 0.0639 -26.2965 -8.1413 9.6401 19 0.909 0.6551 -0.8521 0.7139 -0.6136 2.5249 0.0666 -0.1134 -0.0152 0.1987 -0.0207 0.1142 -0.0204 -0.0673 -0.1003 0.1125 0.0165 0.0807 0.0947 0.1084 0.0822 -33.1526 -17.7682 19.4289 20 0.9425 1.2192 -0.7788 1.6553 -0.8436 2.0978 -0.0228 0.052 -0.0023 -0.0623 -0.0039 0.1565 0.1779 -0.1338 -0.0569 0.0601 0.0325 0.0074 0.0589 0.013 0.0855 -21.7007 -6.9269 3.5059 21 1.0276 -0.5607 0.5709 1.7586 -0.0801 2.4963 -0.0073 -0.1286 -0.0943 0.158 -0.0579 0.0944 0.157 -0.0803 0.0679 0.071 -0.0184 0.0429 0.1264 0.0567 0.086 3.7226 -21.373 52.8869 22 0.0313 -0.0417 0.0328 0.1063 -0.0312 0.2835 -0.0022 0.0188 -0.0206 -0.0398 0.0128 -0.0379 0.0831 0.0026 -0.0393 0.0971 0.0001 -0.0346 -0.0282 -0.0343 0.0949 5.1983 -23.091 37.8455 23 1.3687 -1.0889 0.017 1.0725 0.7074 2.5116 0.0109 0.0507 -0.1053 -0.0313 -0.0056 0.0496 0.1204 -0.0401 0.0001 0.1568 -0.0869 -0.0909 0.1388 -0.053 0.0943 3.5191 -17.7518 17.0363 24 0.3243 0.0114 0.4397 0.3507 0.3538 4.6173 0.0068 -0.0227 -0.1038 0.0743 0.089 -0.0343 0.2802 0.1234 -0.2145 0.133 0.0232 -0.0538 0.1045 -0.0447 0.1004 8.2948 -23.4786 31.8982 25 3.5996 -0.7953 -0.8639 2.1593 0.036 0.2202 -0.0067 -0.3361 0.1124 0.0943 0.0237 -0.1789 -0.0003 0.124 -0.0269 0.0318 0.0321 -0.0109 0.1359 0.0066 0.055 7.9291 -14.776 51.3938 26 0.2233 0.2004 0.0856 0.7558 -0.1794 0.284 0.0702 0.2264 -0.0474 -0.2139 0.1223 0.0314 0.0266 0.1121 0.1798 0.0653 0.0185 -0.0061 0.1557 -0.0347 0.033 8.581 -15.9285 32.3294 27 0.4822 -0.0242 0.0314 0.0853 -0.1381 0.2262 -0.0251 -0.0664 -0.003 0.0197 0.0338 -0.0397 -0.0198 -0.029 0.0122 0.0241 0.0275 0.0101 -0.036 -0.0458 0.0828 -3.9477 -13.062 49.4132 28 1.2759 0.4173 -1.0153 2.3643 -2.4557 2.8385 0.0681 0.192 0.0712 -0.2254 0.0737 0.0624 -0.0985 -0.0034 -0.0465 0.149 -0.0377 -0.0005 0.1603 0.0432 0.0598 -2.1576 -7.9804 28.4903 29 0.4623 0.1299 -0.4229 0.1975 0.1237 0.751 0.0355 0.1172 0.0809 -0.0695 0.1028 0.0369 -0.0201 -0.0555 0.1855 0.0306 -0.0327 -0.007 0.1151 0.0375 0.0016 -3.0424 -12.4729 26.9637 30 1.039 -0.527 -0.0527 5.2534 -2.1524 1.306 -0.0365 -0.0103 -0.0851 -0.0416 0.0287 0.032 0.0555 -0.029 -0.1051 0.1968 -0.0425 -0.0145 0.0561 0.0226 0.0857 -2.1445 -25.8567 42.7788 31 0.4696 0.1731 -0.0436 0.4154 -0.2412 0.6691 0.0236 0.1491 -0.0095 -0.1176 0.0735 0.0675 0.0568 -0.0219 0.0465 0.0808 -0.0369 -0.0258 0.0699 -0.0243 0.0293 0.807 -18.1995 31.0804 32 1.2732 0.55 1.7853 1.0683 1.2813 3.4554 -0.0722 0.1793 0.0318 -0.0886 0.0562 -0.0281 -0.1169 0.1677 0.0409 0.1596 -0.1047 -0.0345 0.2805 -0.0195 0.0615 9.2033 -6.6548 15.2051 33 0.8984 0.219 -0.4828 0.1651 -0.3322 0.6649 0.0245 0.0688 0.0415 0.0598 -0.0235 -0.0313 -0.1796 0.1543 -0.1152 0.1327 -0.0865 -0.0059 0.1781 -0.0027 0.0271 8.6005 -3.9879 31.6011 34 1.3726 1.1306 -1.5637 2.0269 -0.993 2.0133 -0.0197 0.1886 0.054 -0.1201 0.0634 0.1024 -0.0734 -0.3065 -0.0318 0.1099 0.0292 0.0197 0.1213 0.0098 0.0619 -9.4857 -4.7766 39.8536 35 5.9546 -0.7692 -2.4062 2.5477 -0.1863 3.1693 0.0788 0.0248 0.1676 -0.0629 -0.0635 -0.1862 -0.0855 0.0807 -0.0654 0.2248 -0.1373 0.0318 0.1363 0.0395 0.0797 10.2675 -0.8617 27.7674 36 0.4049 0.2208 -0.6873 1.0307 -0.2099 1.218 0.0441 0.107 0.1496 -0.1346 -0.0004 -0.1507 -0.2499 0.0882 -0.097 0.0918 -0.0709 0.0303 0.1922 0.1368 0.1327 1.7575 -2.9434 30.047 37 0.701 0.5324 -0.1173 1.9283 0.0502 0.3038 -0.0592 0.0668 -0.0136 0.015 0.0459 0.102 0.0449 -0.195 -0.0123 0.1036 -0.0031 0.0222 0.0927 0.018 0.0503 -10.4027 -11.0333 46.7668 38 6.455 -3.9959 -4.0416 2.8799 2.8242 3.2715 -0.0246 -0.039 0.1524 0.0603 -0.0271 -0.0338 -0.125 0.1099 -0.0448 0.1268 -0.0368 0.0402 0.0849 0.0012 0.0857 9.4758 -6.6379 39.1879 39 2.6132 -0.4656 -1.1264 1.5909 0.0535 2.2149 0.102 0.2745 -0.0221 -0.2482 0.0344 -0.1174 0.0029 0.1039 -0.0774 0.1066 0.0315 0.0547 0.2366 -0.021 0.0831 16.3754 -16.4916 32.9703 40 1.1203 -0.0218 -0.9218 0.4458 0.0801 2.149 0.0614 0.0206 0.2407 0.1096 -0.0034 0.1832 -0.3302 -0.044 0.0385 0.1704 -0.0204 -0.0002 0.0808 -0.0495 0.2632 3.6818 -3.1612 46.7644
Refinement TLS group Show large table (4 x 40) Hide large table ID Refine-ID Refine TLS-ID Selection details 1 X-RAY DIFFRACTION 1 (chain A and resid 6:11)2 X-RAY DIFFRACTION 2 (chain A and resid 12:25)3 X-RAY DIFFRACTION 3 (chain A and resid 26:30)4 X-RAY DIFFRACTION 4 (chain A and resid 31:38)5 X-RAY DIFFRACTION 5 (chain A and resid 39:44)6 X-RAY DIFFRACTION 6 (chain A and resid 45:55)7 X-RAY DIFFRACTION 7 (chain A and resid 56:72)8 X-RAY DIFFRACTION 8 (chain A and resid 73:91)9 X-RAY DIFFRACTION 9 (chain A and resid 92:101)10 X-RAY DIFFRACTION 10 (chain A and resid 102:109)11 X-RAY DIFFRACTION 11 (chain A and resid 110:115)12 X-RAY DIFFRACTION 12 (chain A and resid 116:128)13 X-RAY DIFFRACTION 13 (chain A and resid 129:138)14 X-RAY DIFFRACTION 14 (chain A and resid 139:146)15 X-RAY DIFFRACTION 15 (chain A and resid 147:167)16 X-RAY DIFFRACTION 16 (chain A and resid 168:181)17 X-RAY DIFFRACTION 17 (chain A and resid 182:195)18 X-RAY DIFFRACTION 18 (chain A and
 Open data
Open data Basic information
Basic information Components
Components Keywords
Keywords Function and homology information
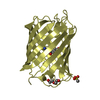
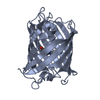
Function and homology information Discosoma sp. LW-2004 (sea anemone)
Discosoma sp. LW-2004 (sea anemone) X-RAY DIFFRACTION /
X-RAY DIFFRACTION /  SYNCHROTRON /
SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2 Å
MOLECULAR REPLACEMENT / Resolution: 2 Å  Authors
Authors Citation
Citation Journal: Plos One / Year: 2012
Journal: Plos One / Year: 2012 Structure visualization
Structure visualization Molmil
Molmil Jmol/JSmol
Jmol/JSmol Downloads & links
Downloads & links Download
Download 4h3m.cif.gz
4h3m.cif.gz PDBx/mmCIF format
PDBx/mmCIF format pdb4h3m.ent.gz
pdb4h3m.ent.gz PDB format
PDB format 4h3m.json.gz
4h3m.json.gz PDBx/mmJSON format
PDBx/mmJSON format Other downloads
Other downloads https://data.pdbj.org/pub/pdb/validation_reports/h3/4h3m
https://data.pdbj.org/pub/pdb/validation_reports/h3/4h3m ftp://data.pdbj.org/pub/pdb/validation_reports/h3/4h3m
ftp://data.pdbj.org/pub/pdb/validation_reports/h3/4h3m


 Links
Links Assembly
Assembly


 Components
Components Discosoma sp. LW-2004 (sea anemone) / Production host:
Discosoma sp. LW-2004 (sea anemone) / Production host: 
 X-RAY DIFFRACTION / Number of used crystals: 1
X-RAY DIFFRACTION / Number of used crystals: 1  Sample preparation
Sample preparation SYNCHROTRON / Site:
SYNCHROTRON / Site:  SSRL
SSRL  / Beamline: BL12-2 / Wavelength: 1.5418, 1.5621, 1.7321
/ Beamline: BL12-2 / Wavelength: 1.5418, 1.5621, 1.7321 Processing
Processing MOLECULAR REPLACEMENT
MOLECULAR REPLACEMENT Movie
Movie Controller
Controller











 PDBj
PDBj

