| 登録情報 | データベース: PDB / ID: 4gb0
|
|---|
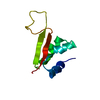
| タイトル | Crystal Structure of the RING domain of RNF168 |
|---|
 要素 要素 | E3 ubiquitin-protein ligase RNF168 |
|---|
 キーワード キーワード | LIGASE / RING domain / E3 ligase / E2 |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
histone H2AK15 ubiquitin ligase activity / histone ubiquitin ligase activity / double-strand break repair via classical nonhomologous end joining / isotype switching / K63-linked polyubiquitin modification-dependent protein binding / response to ionizing radiation / DNA repair-dependent chromatin remodeling / negative regulation of transcription elongation by RNA polymerase II / protein K63-linked ubiquitination / ubiquitin ligase complex ...histone H2AK15 ubiquitin ligase activity / histone ubiquitin ligase activity / double-strand break repair via classical nonhomologous end joining / isotype switching / K63-linked polyubiquitin modification-dependent protein binding / response to ionizing radiation / DNA repair-dependent chromatin remodeling / negative regulation of transcription elongation by RNA polymerase II / protein K63-linked ubiquitination / ubiquitin ligase complex / interstrand cross-link repair / SUMOylation of DNA damage response and repair proteins / nucleosome binding / positive regulation of DNA repair / epigenetic regulation of gene expression / ubiquitin binding / Nonhomologous End-Joining (NHEJ) / G2/M DNA damage checkpoint / RING-type E3 ubiquitin transferase / double-strand break repair via nonhomologous end joining / ubiquitin-protein transferase activity / Recruitment and ATM-mediated phosphorylation of repair and signaling proteins at DNA double strand breaks / site of double-strand break / double-strand break repair / Processing of DNA double-strand break ends / ubiquitin-dependent protein catabolic process / histone binding / protein ubiquitination / DNA damage response / chromatin binding / protein-containing complex / zinc ion binding / nucleoplasm / nucleus / cytosol類似検索 - 分子機能 E3 ubiquitin-protein ligase RNF168 / : / Prokaryotic RING finger family 4 / Zinc/RING finger domain, C3HC4 (zinc finger) / Herpes Virus-1 / Ring finger / Zinc finger RING-type profile. / Zinc finger, RING-type / Zinc finger, RING/FYVE/PHD-type / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 MALONATE ION / E3 ubiquitin-protein ligase RNF168類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  単波長異常分散 / 解像度: 2.6 Å 単波長異常分散 / 解像度: 2.6 Å |
|---|
 データ登録者 データ登録者 | Zhang, X.Q. / Wang, C.L. / Zang, J.Y. |
|---|
 引用 引用 |  ジャーナル: Cell Cycle / 年: 2013 ジャーナル: Cell Cycle / 年: 2013
タイトル: Structural basis for role of ring finger protein RNF168 RING domain
著者: Zhang, X.Q. / Chen, J. / Wu, M. / Wu, H. / Arokiaraj, A.W. / Wang, C.L. / Zhang, W. / Tao, Y. / Huen, M.S. / Zang, J.Y. |
|---|
| 履歴 | | 登録 | 2012年7月26日 | 登録サイト: RCSB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2013年7月10日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年3月20日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr2_auth_seq_id / _pdbx_struct_conn_angle.ptnr2_label_asym_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  単波長異常分散 / 解像度: 2.6 Å
単波長異常分散 / 解像度: 2.6 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Cell Cycle / 年: 2013
ジャーナル: Cell Cycle / 年: 2013 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4gb0.cif.gz
4gb0.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4gb0.ent.gz
pdb4gb0.ent.gz PDB形式
PDB形式 4gb0.json.gz
4gb0.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4gb0_validation.pdf.gz
4gb0_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4gb0_full_validation.pdf.gz
4gb0_full_validation.pdf.gz 4gb0_validation.xml.gz
4gb0_validation.xml.gz 4gb0_validation.cif.gz
4gb0_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/gb/4gb0
https://data.pdbj.org/pub/pdb/validation_reports/gb/4gb0 ftp://data.pdbj.org/pub/pdb/validation_reports/gb/4gb0
ftp://data.pdbj.org/pub/pdb/validation_reports/gb/4gb0 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 遺伝子: RNF168 / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: RNF168 / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SSRF
SSRF  / ビームライン: BL17U / 波長: 0.9792 Å
/ ビームライン: BL17U / 波長: 0.9792 Å 解析
解析 単波長異常分散 / 解像度: 2.6→35.47 Å / Cor.coef. Fo:Fc: 0.938 / Cor.coef. Fo:Fc free: 0.914 / 交差検証法: THROUGHOUT / ESU R: 0.437 / ESU R Free: 0.307 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
単波長異常分散 / 解像度: 2.6→35.47 Å / Cor.coef. Fo:Fc: 0.938 / Cor.coef. Fo:Fc free: 0.914 / 交差検証法: THROUGHOUT / ESU R: 0.437 / ESU R Free: 0.307 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj









