| 登録情報 | データベース: PDB / ID: 4f8a
|
|---|
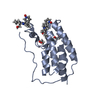
| タイトル | Cyclic nucleotide binding-homology domain from mouse EAG1 potassium channel |
|---|
 要素 要素 | Potassium voltage-gated channel subfamily H member 1 |
|---|
 キーワード キーワード | MEMBRANE PROTEIN / TRANSPORT PROTEIN / probable regulatory domain of potassium channel |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Voltage gated Potassium channels / potassium channel complex / regulation of presynaptic cytosolic calcium ion concentration / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / delayed rectifier potassium channel activity / nuclear inner membrane / phosphatidylinositol bisphosphate binding / regulation of synaptic vesicle exocytosis / startle response / parallel fiber to Purkinje cell synapse ...Voltage gated Potassium channels / potassium channel complex / regulation of presynaptic cytosolic calcium ion concentration / voltage-gated monoatomic ion channel activity involved in regulation of presynaptic membrane potential / delayed rectifier potassium channel activity / nuclear inner membrane / phosphatidylinositol bisphosphate binding / regulation of synaptic vesicle exocytosis / startle response / parallel fiber to Purkinje cell synapse / axolemma / 14-3-3 protein binding / voltage-gated potassium channel complex / potassium ion transmembrane transport / cellular response to calcium ion / regulation of membrane potential / potassium ion transport / postsynaptic density membrane / regulation of cell population proliferation / presynaptic membrane / early endosome membrane / monoatomic ion transmembrane transport / perikaryon / transmembrane transporter binding / calmodulin binding / axon / neuronal cell body / dendrite / protein kinase binding / protein-containing complex binding / perinuclear region of cytoplasm / cell surface / identical protein binding / plasma membrane類似検索 - 分子機能 Potassium channel, voltage-dependent, EAG / : / Potassium channel, voltage-dependent, EAG/ELK/ERG / PAS domain / PAS-associated, C-terminal / PAC domain profile. / PAC motif / Motif C-terminal to PAS motifs (likely to contribute to PAS structural domain) / Cyclic nucleotide-monophosphate binding domain / Cyclic nucleotide-binding domain ...Potassium channel, voltage-dependent, EAG / : / Potassium channel, voltage-dependent, EAG/ELK/ERG / PAS domain / PAS-associated, C-terminal / PAC domain profile. / PAC motif / Motif C-terminal to PAS motifs (likely to contribute to PAS structural domain) / Cyclic nucleotide-monophosphate binding domain / Cyclic nucleotide-binding domain / cAMP/cGMP binding motif profile. / Cyclic nucleotide-binding domain / Cyclic nucleotide-binding domain superfamily / Jelly Rolls / PAS repeat profile. / PAS domain / RmlC-like jelly roll fold / PAS domain superfamily / Ion transport domain / Ion transport protein / Jelly Rolls / Sandwich / Mainly Beta類似検索 - ドメイン・相同性 Voltage-gated delayed rectifier potassium channel KCNH1類似検索 - 構成要素 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.2 Å 分子置換 / 解像度: 2.2 Å |
|---|
 データ登録者 データ登録者 | Marques-Carvalho, M.J. / Sahoo, N. / Muskett, F.W. / Vieira-Pires, R.S. / Gabant, G. / Cadene, M. / Schonherr, R. / Morais-Cabral, J.H. |
|---|
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2012タイトル ジャーナル: J.Mol.Biol. / 年: 2012タイトル: Structural, Biochemical, and Functional Characterization of the Cyclic Nucleotide Binding Homology Domain from the Mouse EAG1 Potassium Channel. 著者: Marques-Carvalho, M.J. / Sahoo, N. / Muskett, F.W. / Vieira-Pires, R.S. / Gabant, G. / Cadene, M. / Schonherr, R. / Morais-Cabral, J.H.#1: ジャーナル: Acta Crystallogr.,Sect.F / 年: 2012タイトル: Crystallization and preliminary X-ray crystallographic characterization of a cyclic nucleotide-binding homology domain from the mouse EAG potassium channel. 著者: Marques-Carvalho, M.J. / Morais-Cabral, J.H. |
|---|
| 履歴 | | 登録 | 2012年5月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2012年7月11日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2012年10月17日 | Group: Database references |
|---|
| 改定 1.2 | 2024年2月28日 | Group: Data collection / Database references
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.2 Å
分子置換 / 解像度: 2.2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 2012
ジャーナル: J.Mol.Biol. / 年: 2012 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4f8a.cif.gz
4f8a.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4f8a.ent.gz
pdb4f8a.ent.gz PDB形式
PDB形式 4f8a.json.gz
4f8a.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/f8/4f8a
https://data.pdbj.org/pub/pdb/validation_reports/f8/4f8a ftp://data.pdbj.org/pub/pdb/validation_reports/f8/4f8a
ftp://data.pdbj.org/pub/pdb/validation_reports/f8/4f8a リンク
リンク 集合体
集合体
 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ESRF
ESRF  / ビームライン: ID14-4 / 波長: 0.9765 Å
/ ビームライン: ID14-4 / 波長: 0.9765 Å 解析
解析 分子置換 / 解像度: 2.2→52.261 Å / Occupancy max: 1 / Occupancy min: 0 / FOM work R set: 0.8415 / SU ML: 0.45 / σ(F): 1.37 / 位相誤差: 22.04 / 立体化学のターゲット値: Engh & Huber
分子置換 / 解像度: 2.2→52.261 Å / Occupancy max: 1 / Occupancy min: 0 / FOM work R set: 0.8415 / SU ML: 0.45 / σ(F): 1.37 / 位相誤差: 22.04 / 立体化学のターゲット値: Engh & Huber ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj







