[English] 日本語
 Yorodumi
Yorodumi- PDB-4e4u: Crystal structure of a putative Mandelate racemase/Muconate lacto... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 4e4u | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of a putative Mandelate racemase/Muconate lactonizing enzyme (Target PSI-200780) from Burkholderia SAR-1 | ||||||
 Components Components | Mandalate racemase/muconate lactonizing enzyme | ||||||
 Keywords Keywords | ISOMERASE / Racemase / Mandelate racemase / aldolase / Structural genomics / PSI-Biology / New York Structural Genomics Research Consortium / NYSGRC | ||||||
| Function / homology | Enolase-like C-terminal domain / Enolase-like, N-terminal domain / Enolase-like; domain 1 / TIM Barrel / Alpha-Beta Barrel / 2-Layer Sandwich / Alpha Beta / (2R,3S)-1,4-DIMERCAPTOBUTANE-2,3-DIOL Function and homology information Function and homology information | ||||||
| Biological species | unidentified (others) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.35 Å MOLECULAR REPLACEMENT / Resolution: 1.35 Å | ||||||
 Authors Authors | Kumar, P.R. / Bonanno, J. / Chowdhury, S. / Foti, R. / Gizzi, A. / Hammonds, J. / Hillerich, B. / Matikainen, B. / Seidel, R. / Toro, R. ...Kumar, P.R. / Bonanno, J. / Chowdhury, S. / Foti, R. / Gizzi, A. / Hammonds, J. / Hillerich, B. / Matikainen, B. / Seidel, R. / Toro, R. / Zencheck, W. / Almo, S.C. / New York Structural Genomics Research Consortium (NYSGRC) | ||||||
 Citation Citation |  Journal: to be published Journal: to be publishedTitle: Crystal structure of a putative MR/ML enzyme from Burkholderia SAR-1 Authors: Kumar, P.R. / Bonanno, J. / Chowdhury, S. / Foti, R. / Gizzi, A. / Hammonds, J. / Hillerich, B. / Matikainen, B. / Seidel, R. / Toro, R. / Zencheck, W. / Almo, S.C. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  4e4u.cif.gz 4e4u.cif.gz | 187.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb4e4u.ent.gz pdb4e4u.ent.gz | 149.5 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  4e4u.json.gz 4e4u.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/e4/4e4u https://data.pdbj.org/pub/pdb/validation_reports/e4/4e4u ftp://data.pdbj.org/pub/pdb/validation_reports/e4/4e4u ftp://data.pdbj.org/pub/pdb/validation_reports/e4/4e4u | HTTPS FTP |
|---|
-Related structure data
| Similar structure data | |
|---|---|
| Other databases |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | x 8
| ||||||||||||||||||
| Unit cell |
| ||||||||||||||||||
| Components on special symmetry positions |
| ||||||||||||||||||
| Details | The biological assembly is an octamer generated by crystallographic symmetry operations |
- Components
Components
| #1: Protein | Mass: 45648.180 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Details: from Sargasso sea / Source: (gene. exp.) unidentified (others) / Production host:  |
|---|---|
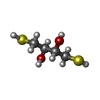
| #2: Chemical | ChemComp-DTU / ( |
| #3: Water | ChemComp-HOH / |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.34 Å3/Da / Density % sol: 47.35 % |
|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, sitting drop / pH: 4.5 Details: Protein (20 mM Hepes, pH 7.5, 150 mM NaCl, 10% glycerol; Reservoir (2.5M NaCl, Sodium Acetate 4.5, 0.2M Li2SO4- Wizard II #38); Cryoprotection (LI2SO4), Sitting Drop Vapor Diffusion, ...Details: Protein (20 mM Hepes, pH 7.5, 150 mM NaCl, 10% glycerol; Reservoir (2.5M NaCl, Sodium Acetate 4.5, 0.2M Li2SO4- Wizard II #38); Cryoprotection (LI2SO4), Sitting Drop Vapor Diffusion, temperature 298K, VAPOR DIFFUSION, SITTING DROP |
-Data collection
| Diffraction | Mean temperature: 100 K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  NSLS NSLS  / Beamline: X29A / Wavelength: 1.075 Å / Beamline: X29A / Wavelength: 1.075 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: ADSC QUANTUM 315 / Detector: CCD / Date: Apr 23, 2011 / Details: MIRRORS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Monochromator: GRAPHITE / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 1.075 Å / Relative weight: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Highest resolution: 1.35 Å / Num. obs: 181393 / % possible obs: 99.9 % / Observed criterion σ(I): -3 / Biso Wilson estimate: 21.261 Å2 / Rmerge(I) obs: 0.089 / Net I/σ(I): 16.05 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell | Rmerge(I) obs: 0.011 / Diffraction-ID: 1
|
- Processing
Processing
| Software |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT / Resolution: 1.35→37.641 Å / Occupancy max: 1 / Occupancy min: 0.23 / FOM work R set: 0.934 / SU ML: 0.16 / σ(F): 1.34 / Phase error: 12.4 / Stereochemistry target values: ML MOLECULAR REPLACEMENT / Resolution: 1.35→37.641 Å / Occupancy max: 1 / Occupancy min: 0.23 / FOM work R set: 0.934 / SU ML: 0.16 / σ(F): 1.34 / Phase error: 12.4 / Stereochemistry target values: ML
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.98 Å / VDW probe radii: 1.2 Å / Solvent model: FLAT BULK SOLVENT MODEL / Bsol: 44.327 Å2 / ksol: 0.393 e/Å3 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 59.07 Å2 / Biso mean: 17.8719 Å2 / Biso min: 5.95 Å2
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.35→37.641 Å
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 30
|
 Movie
Movie Controller
Controller










 PDBj
PDBj