| 登録情報 | データベース: PDB / ID: 4dko
|
|---|
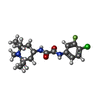
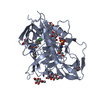
| タイトル | Crystal structure of clade A/E 93TH057 HIV-1 gp120 core in complex with TS-II-224 |
|---|
 要素 要素 | HIV-1 gp120 core |
|---|
 キーワード キーワード | VIRAL PROTEIN/INHIBITOR / HIV-1 gp120 / clade A/E / CD4 mimic / TS-II-224 / VIRAL PROTEIN-INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope ...positive regulation of plasma membrane raft polarization / positive regulation of receptor clustering / positive regulation of establishment of T cell polarity / host cell endosome membrane / clathrin-dependent endocytosis of virus by host cell / viral protein processing / fusion of virus membrane with host plasma membrane / virus-mediated perturbation of host defense response / fusion of virus membrane with host endosome membrane / viral envelope / virion attachment to host cell / apoptotic process / host cell plasma membrane / structural molecule activity / virion membrane / plasma membrane類似検索 - 分子機能 HIV Envelope Protein Gp120; Chain G / Human immunodeficiency virus 1, Gp160, envelope glycoprotein / Envelope glycoprotein Gp160 / Retroviral envelope protein / Retroviral envelope protein GP41-like / Gp120 core superfamily / Envelope glycoprotein GP120 / Human immunodeficiency virus 1, envelope glycoprotein Gp120 / Beta Complex / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 (ヒト免疫不全ウイルス) HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 (ヒト免疫不全ウイルス) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.981 Å 分子置換 / 解像度: 1.981 Å |
|---|
 データ登録者 データ登録者 | Kwon, Y.D. / LaLonde, J.M. / Jones, D.M. / Sun, A.W. / Courter, J.R. / Soeta, T. / Kobayashi, T. / Princiotto, A.M. / Wu, X. / Mascola, J. ...Kwon, Y.D. / LaLonde, J.M. / Jones, D.M. / Sun, A.W. / Courter, J.R. / Soeta, T. / Kobayashi, T. / Princiotto, A.M. / Wu, X. / Mascola, J. / Schon, A. / Freire, E. / Sodroski, J. / Madani, N. / Smith III, A.B. / Kwong, P.D. |
|---|
 引用 引用 |  ジャーナル: J.Med.Chem. / 年: 2012 ジャーナル: J.Med.Chem. / 年: 2012
タイトル: Structure-Based Design, Synthesis, and Characterization of Dual Hotspot Small-Molecule HIV-1 Entry Inhibitors.
著者: Lalonde, J.M. / Kwon, Y.D. / Jones, D.M. / Sun, A.W. / Courter, J.R. / Soeta, T. / Kobayashi, T. / Princiotto, A.M. / Wu, X. / Schon, A. / Freire, E. / Kwong, P.D. / Mascola, J.R. / Sodroski, ...著者: Lalonde, J.M. / Kwon, Y.D. / Jones, D.M. / Sun, A.W. / Courter, J.R. / Soeta, T. / Kobayashi, T. / Princiotto, A.M. / Wu, X. / Schon, A. / Freire, E. / Kwong, P.D. / Mascola, J.R. / Sodroski, J. / Madani, N. / Smith, A.B. |
|---|
| 履歴 | | 登録 | 2012年2月3日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2012年5月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2012年5月9日 | Group: Source and taxonomy |
|---|
| 改定 1.2 | 2012年6月20日 | Group: Database references |
|---|
| 改定 1.3 | 2020年7月29日 | Group: Data collection / Derived calculations / Structure summary
カテゴリ: chem_comp / entity ...chem_comp / entity / pdbx_chem_comp_identifier / pdbx_entity_nonpoly / struct_conn / struct_site / struct_site_gen
Item: _chem_comp.name / _chem_comp.type ..._chem_comp.name / _chem_comp.type / _entity.pdbx_description / _pdbx_entity_nonpoly.name / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.pdbx_role / _struct_conn.ptnr1_auth_asym_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_asym_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id
解説: Carbohydrate remediation / Provider: repository / タイプ: Remediation |
|---|
| 改定 1.4 | 2021年5月19日 | Group: Source and taxonomy / Structure summary / カテゴリ: chem_comp / entity_src_gen
Item: _chem_comp.pdbx_synonyms / _entity_src_gen.host_org_common_name ..._chem_comp.pdbx_synonyms / _entity_src_gen.host_org_common_name / _entity_src_gen.pdbx_host_org_cell_line / _entity_src_gen.pdbx_host_org_strain |
|---|
| 改定 1.5 | 2023年9月13日 | Group: Data collection / Database references / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 (ヒト免疫不全ウイルス)
HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 (ヒト免疫不全ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.981 Å
分子置換 / 解像度: 1.981 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Med.Chem. / 年: 2012
ジャーナル: J.Med.Chem. / 年: 2012 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 4dko.cif.gz
4dko.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb4dko.ent.gz
pdb4dko.ent.gz PDB形式
PDB形式 4dko.json.gz
4dko.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 4dko_validation.pdf.gz
4dko_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 4dko_full_validation.pdf.gz
4dko_full_validation.pdf.gz 4dko_validation.xml.gz
4dko_validation.xml.gz 4dko_validation.cif.gz
4dko_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/dk/4dko
https://data.pdbj.org/pub/pdb/validation_reports/dk/4dko ftp://data.pdbj.org/pub/pdb/validation_reports/dk/4dko
ftp://data.pdbj.org/pub/pdb/validation_reports/dk/4dko リンク
リンク 集合体
集合体


 要素
要素
 HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 (ヒト免疫不全ウイルス)
HUMAN IMMUNODEFICIENCY VIRUS TYPE 1 (ヒト免疫不全ウイルス) Homo sapiens (ヒト) / 参照: UniProt: Q0ED31*PLUS
Homo sapiens (ヒト) / 参照: UniProt: Q0ED31*PLUS X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  APS
APS  / ビームライン: 22-ID / 波長: 1 Å
/ ビームライン: 22-ID / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー


























 PDBj
PDBj