+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4bvh | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CRYSTAL STRUCTURE OF HUMAN SIRT3 IN COMPLEX WITH THE INHIBITOR EX-527 AND 2'-O-ACETYL-ADP-RIBOSE | ||||||
 要素 要素 | NAD-DEPENDENT PROTEIN DEACETYLASE SIRTUIN-3, MITOCHONDRIAL | ||||||
 キーワード キーワード | HYDROLASE / INHIBITOR COMPLEX / EX-527 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報positive regulation of superoxide dismutase activity / positive regulation of catalase activity / NAD-dependent protein lysine delactylase activity / positive regulation of ceramide biosynthetic process / Maturation of TCA enzymes and regulation of TCA cycle / peptidyl-lysine deacetylation / NAD-dependent protein lysine deacetylase activity / protein acetyllysine N-acetyltransferase / protein deacetylation / histone deacetylase activity, NAD-dependent ...positive regulation of superoxide dismutase activity / positive regulation of catalase activity / NAD-dependent protein lysine delactylase activity / positive regulation of ceramide biosynthetic process / Maturation of TCA enzymes and regulation of TCA cycle / peptidyl-lysine deacetylation / NAD-dependent protein lysine deacetylase activity / protein acetyllysine N-acetyltransferase / protein deacetylation / histone deacetylase activity, NAD-dependent / positive regulation of oxidative phosphorylation / Regulation of FOXO transcriptional activity by acetylation / protein lysine deacetylase activity / cellular response to stress / negative regulation of reactive oxygen species metabolic process / NAD+ binding / Mitochondrial unfolded protein response (UPRmt) / FOXO-mediated transcription of oxidative stress, metabolic and neuronal genes / 転移酵素; アシル基を移すもの; アミノアシル基以外のアシル基を移すもの / aerobic respiration / Transcriptional activation of mitochondrial biogenesis / positive regulation of insulin secretion / negative regulation of ERK1 and ERK2 cascade / sequence-specific DNA binding / mitochondrial matrix / enzyme binding / protein-containing complex / mitochondrion / zinc ion binding / nucleoplasm / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.9 Å 分子置換 / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Gertz, M. / Weyand, M. / Steegborn, C. | ||||||
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2013 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2013タイトル: Ex-527 Inhibits Sirtuins by Exploiting Their Unique Nad+-Dependent Deacetylation Mechanism 著者: Gertz, M. / Fischer, F. / Nguyen, G.T.T. / Lakshminarasimhan, M. / Schutkowski, M. / Weyand, M. / Steegborn, C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4bvh.cif.gz 4bvh.cif.gz | 192.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4bvh.ent.gz pdb4bvh.ent.gz | 150.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4bvh.json.gz 4bvh.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/bv/4bvh https://data.pdbj.org/pub/pdb/validation_reports/bv/4bvh ftp://data.pdbj.org/pub/pdb/validation_reports/bv/4bvh ftp://data.pdbj.org/pub/pdb/validation_reports/bv/4bvh | HTTPS FTP |
|---|
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||||||
| 2 | 
| ||||||||||||
| 3 | 
| ||||||||||||
| 単位格子 |
| ||||||||||||
| 非結晶学的対称性 (NCS) | NCS oper:
|
- 要素
要素
-タンパク質 , 1種, 3分子 ABC
| #1: タンパク質 | 分子量: 31484.219 Da / 分子数: 3 / 断片: RESIDUES 116-399 / 由来タイプ: 組換発現 / 由来: (組換発現)  HOMO SAPIENS (ヒト) / プラスミド: PVFT3S / 発現宿主: HOMO SAPIENS (ヒト) / プラスミド: PVFT3S / 発現宿主:  参照: UniProt: Q9NTG7, 加水分解酵素; ペプチド以外のCN結合加水分解酵素; 鎖状アミドに作用 |
|---|
-非ポリマー , 9種, 506分子 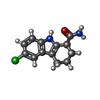
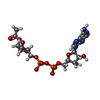



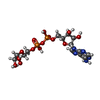











| #2: 化合物 | | #3: 化合物 | #4: 化合物 | #5: 化合物 | ChemComp-NA / | #6: 化合物 | #7: 化合物 | ChemComp-AR6 / [( | #8: 化合物 | ChemComp-EDO / #9: 化合物 | ChemComp-GOL / | #10: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.26 Å3/Da / 溶媒含有率: 45.52 % / 解説: NONE |
|---|---|
| 結晶化 | pH: 7 / 詳細: 20% PEG 6000, 0.1 M HEPES PH7 |
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  BESSY BESSY  / ビームライン: 14.1 / 波長: 0.918 / ビームライン: 14.1 / 波長: 0.918 |
| 検出器 | タイプ: MARMOSAIC 225 mm CCD / 検出器: CCD / 日付: 2012年5月30日 / 詳細: COLLIMATOR |
| 放射 | モノクロメーター: SI(111) MONOCHROMATOR / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.918 Å / 相対比: 1 |
| 反射 | 解像度: 1.9→34.4 Å / Num. obs: 68289 / % possible obs: 99.8 % / Observed criterion σ(I): -3 / 冗長度: 4.7 % / Rmerge(I) obs: 0.08 / Net I/σ(I): 16.3 |
| 反射 シェル | 解像度: 1.9→1.95 Å / 冗長度: 4.8 % / Rmerge(I) obs: 0.49 / Mean I/σ(I) obs: 3.5 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 3GLT 解像度: 1.9→34.44 Å / Cor.coef. Fo:Fc: 0.959 / Cor.coef. Fo:Fc free: 0.927 / SU B: 3.076 / SU ML: 0.092 / 交差検証法: THROUGHOUT / ESU R: 0.147 / ESU R Free: 0.145 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. HYDROGENS HAVE BEEN USED IF PRESENT IN THE INPUT. U VALUES REFINED INDIVIDUALLY
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.2 Å / 溶媒モデル: MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 27.027 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→34.44 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー





















 PDBj
PDBj


















