+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 4b2q | ||||||
|---|---|---|---|---|---|---|---|
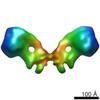
| タイトル | Model of the yeast F1Fo-ATP synthase dimer based on subtomogram average | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | HYDROLASE / SUBTOMOGRAM AVERAGE | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / : / Formation of ATP by chemiosmotic coupling / Cristae formation / Mitochondrial protein degradation / Mitochondrial protein degradation / proton-transporting ATP synthase complex / proton transmembrane transporter activity / proton motive force-driven ATP synthesis / mitochondrial nucleoid ...: / : / Formation of ATP by chemiosmotic coupling / Cristae formation / Mitochondrial protein degradation / Mitochondrial protein degradation / proton-transporting ATP synthase complex / proton transmembrane transporter activity / proton motive force-driven ATP synthesis / mitochondrial nucleoid / : / proton motive force-driven mitochondrial ATP synthesis / : / H+-transporting two-sector ATPase / proton-transporting ATPase activity, rotational mechanism / proton-transporting ATP synthase activity, rotational mechanism / ADP binding / mitochondrial intermembrane space / mitochondrial inner membrane / lipid binding / ATP hydrolysis activity / mitochondrion / ATP binding / identical protein binding / plasma membrane / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |   | ||||||
| 手法 | 電子顕微鏡法 / 電子線トモグラフィー法 / クライオ電子顕微鏡法 / 解像度: 37 Å | ||||||
 データ登録者 データ登録者 | Davies, K.M. / Kuehlbrandt, W. | ||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2012 ジャーナル: Proc Natl Acad Sci U S A / 年: 2012タイトル: Structure of the yeast F1Fo-ATP synthase dimer and its role in shaping the mitochondrial cristae. 著者: Karen M Davies / Claudio Anselmi / Ilka Wittig / José D Faraldo-Gómez / Werner Kühlbrandt /  要旨: We used electron cryotomography of mitochondrial membranes from wild-type and mutant Saccharomyces cerevisiae to investigate the structure and organization of ATP synthase dimers in situ. Subtomogram ...We used electron cryotomography of mitochondrial membranes from wild-type and mutant Saccharomyces cerevisiae to investigate the structure and organization of ATP synthase dimers in situ. Subtomogram averaging of the dimers to 3.7 nm resolution revealed a V-shaped structure of twofold symmetry, with an angle of 86° between monomers. The central and peripheral stalks are well resolved. The monomers interact within the membrane at the base of the peripheral stalks. In wild-type mitochondria ATP synthase dimers are found in rows along the highly curved cristae ridges, and appear to be crucial for membrane morphology. Strains deficient in the dimer-specific subunits e and g or the first transmembrane helix of subunit 4 lack both dimers and lamellar cristae. Instead, cristae are either absent or balloon-shaped, with ATP synthase monomers distributed randomly in the membrane. Computer simulations indicate that isolated dimers induce a plastic deformation in the lipid bilayer, which is partially relieved by their side-by-side association. We propose that the assembly of ATP synthase dimer rows is driven by the reduction in the membrane elastic energy, rather than by direct protein contacts, and that the dimer rows enable the formation of highly curved ridges in mitochondrial cristae. | ||||||
| 履歴 |
| ||||||
| Remark 700 | SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "AA" IN EACH CHAIN ON SHEET RECORDS BELOW ... SHEET DETERMINATION METHOD: DSSP THE SHEETS PRESENTED AS "AA" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 10-STRANDED BARREL THIS IS REPRESENTED BY A 11-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL. THE SHEETS PRESENTED AS "BA" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 10-STRANDED BARREL THIS IS REPRESENTED BY A 11-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL. THE SHEETS PRESENTED AS "CA" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 10-STRANDED BARREL THIS IS REPRESENTED BY A 11-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL. THE SHEETS PRESENTED AS "AA" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 10-STRANDED BARREL THIS IS REPRESENTED BY A 11-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL. THE SHEETS PRESENTED AS "BA" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 10-STRANDED BARREL THIS IS REPRESENTED BY A 11-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL. THE SHEETS PRESENTED AS "CA" IN EACH CHAIN ON SHEET RECORDS BELOW IS ACTUALLY AN 10-STRANDED BARREL THIS IS REPRESENTED BY A 11-STRANDED SHEET IN WHICH THE FIRST AND LAST STRANDS ARE IDENTICAL. |
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  4b2q.cif.gz 4b2q.cif.gz | 1.5 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb4b2q.ent.gz pdb4b2q.ent.gz | 1.2 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  4b2q.json.gz 4b2q.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  4b2q_validation.pdf.gz 4b2q_validation.pdf.gz | 1.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  4b2q_full_validation.pdf.gz 4b2q_full_validation.pdf.gz | 1.6 MB | 表示 | |
| XML形式データ |  4b2q_validation.xml.gz 4b2q_validation.xml.gz | 210.8 KB | 表示 | |
| CIF形式データ |  4b2q_validation.cif.gz 4b2q_validation.cif.gz | 337.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/b2/4b2q https://data.pdbj.org/pub/pdb/validation_reports/b2/4b2q ftp://data.pdbj.org/pub/pdb/validation_reports/b2/4b2q ftp://data.pdbj.org/pub/pdb/validation_reports/b2/4b2q | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
|
|---|---|
| 1 |
|
- 要素
要素
-ATP SYNTHASE SUBUNIT ... , 11種, 44分子 ACacBbDdEFefGgHhIiJKLMNOPQRSjk...
| #1: タンパク質 | 分子量: 52376.539 Da / 分子数: 4 / 断片: RESIDUES 61-545 / 由来タイプ: 天然 / 由来: (天然)  #2: タンパク質 | 分子量: 52447.617 Da / 分子数: 2 / 断片: RESIDUES 60-545 / 由来タイプ: 天然 / 由来: (天然)  #3: タンパク質 | 分子量: 50438.348 Da / 分子数: 2 / 断片: RESIDUES 39-508 / 由来タイプ: 天然 / 由来: (天然)  #4: タンパク質 | 分子量: 50752.641 Da / 分子数: 4 / 断片: RESIDUES 39-511 / 由来タイプ: 天然 / 由来: (天然)  #5: タンパク質 | 分子量: 30657.160 Da / 分子数: 2 / 断片: RESIDUES 34-311 / 由来タイプ: 天然 / 由来: (天然)  #6: タンパク質 | 分子量: 14080.876 Da / 分子数: 2 / 断片: RESIDUES 29-160 / 由来タイプ: 天然 / 由来: (天然)  #7: タンパク質 | 分子量: 6388.076 Da / 分子数: 2 / 断片: RESIDUES 2-60 / 由来タイプ: 天然 / 由来: (天然)  #8: タンパク質 | 分子量: 7762.375 Da / 分子数: 20 / 由来タイプ: 天然 / 由来: (天然)  #9: タンパク質 | 分子量: 15408.768 Da / 分子数: 2 / 断片: RESIDUES 121-249 / 由来タイプ: 天然 / 由来: (天然)  #10: タンパク質 | 分子量: 13768.688 Da / 分子数: 2 / 断片: RESIDUES 5-124 / 由来タイプ: 天然 / 由来: (天然)  #12: タンパク質 | 分子量: 13240.513 Da / 分子数: 2 / 断片: RESIDUES 24-143 / 由来タイプ: 天然 / 由来: (天然)  |
|---|
-タンパク質 , 1種, 2分子 Vv
| #11: タンパク質 | 分子量: 7829.806 Da / 分子数: 2 / 断片: RESIDUES 36-101 / 由来タイプ: 天然 / 由来: (天然)  |
|---|
-非ポリマー , 3種, 20分子 




| #13: 化合物 | ChemComp-ATP / #14: 化合物 | ChemComp-MG / #15: 化合物 | ChemComp-ADP / |
|---|
-実験情報
-実験
| 実験 | 手法: 電子顕微鏡法 |
|---|---|
| EM実験 | 試料の集合状態: CELL / 3次元再構成法: 電子線トモグラフィー法 |
- 試料調製
試料調製
| 構成要素 | 名称: ATP SYNTHASE DIMER FROM MITOCHONDRIA OF SACCHAROMYCES CEREVISIAE タイプ: CELL |
|---|---|
| 緩衝液 | 名称: 250MM TREHALOSE 10NM TRIS- HCL PH7.4 / pH: 7.4 / 詳細: 250MM TREHALOSE 10NM TRIS- HCL PH7.4 |
| 試料 | 包埋: NO / シャドウイング: NO / 染色: NO / 凍結: YES |
| 試料支持 | 詳細: HOLEY CARBON |
| 急速凍結 | 装置: HOMEMADE PLUNGER / 凍結剤: ETHANE 詳細: VITRIFICATION 1 -- CRYOGEN- ETHANE, TEMPERATURE- 100, INSTRUMENT- HOMEMADE PLUNGER, METHOD- SINGLE SIDE MANUAL BLOTTING FOR 5 SECONDS., |
- 電子顕微鏡撮影
電子顕微鏡撮影
| 実験機器 |  モデル: Tecnai Polara / 画像提供: FEI Company |
|---|---|
| 顕微鏡 | モデル: FEI POLARA 300 / 日付: 2009年3月11日 |
| 電子銃 | 電子線源:  FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM FIELD EMISSION GUN / 加速電圧: 300 kV / 照射モード: FLOOD BEAM |
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 41000 X / 倍率(補正後): 24500 X / 最大 デフォーカス(公称値): 7500 nm / 最小 デフォーカス(公称値): 6500 nm / Cs: 2 mm |
| 試料ホルダ | 温度: 80 K / 傾斜角・最大: 60 ° / 傾斜角・最小: -50 ° |
| 撮影 | 電子線照射量: 160 e/Å2 フィルム・検出器のモデル: GATAN ULTRASCAN 1000 (2k x 2k) |
| 画像スキャン | デジタル画像の数: 75 |
| 放射波長 | 相対比: 1 |
- 解析
解析
| EMソフトウェア |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 対称性 | 点対称性: C2 (2回回転対称) | ||||||||||||
| 3次元再構成 | 手法: WEIGHTED BACK-PROJECTION / 解像度: 37 Å / 粒子像の数: 121 / ピクセルサイズ(実測値): 5.76 Å 詳細: MODEL BASED ON PDBS 2WPD,2CLY AND 2BO5. SUBMISSION BASED ON EXPERIMENTAL DATA FROM EMDB EMD-2161. (DEPOSITION ID: 10941). 対称性のタイプ: POINT | ||||||||||||
| 原子モデル構築 | プロトコル: RIGID BODY FIT / 空間: REAL / 詳細: METHOD--RIGID BODY REFINEMENT PROTOCOL--X-RAY | ||||||||||||
| 原子モデル構築 | PDB-ID: 2WPD Accession code: 2WPD / Source name: PDB / タイプ: experimental model | ||||||||||||
| 精密化 | 最高解像度: 37 Å | ||||||||||||
| 精密化ステップ | サイクル: LAST / 最高解像度: 37 Å
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj





