[English] 日本語
 Yorodumi
Yorodumi- PDB-3vet: Crystal structure of the O-carbamoyltransferase TobZ in complex w... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 3vet | ||||||
|---|---|---|---|---|---|---|---|
| Title | Crystal structure of the O-carbamoyltransferase TobZ in complex with Tobramycin, carbamoyl phosphate and ADP | ||||||
 Components Components | O-carbamoyltransferase TobZ | ||||||
 Keywords Keywords | TRANSFERASE/ANTIBIOTIC / antibiotic biosynthesis / substrate assisted catalysis / substrate channeling / adenylation / structural enzymology / enzyme evolution / TRANSFERASE-ANTIBIOTIC complex | ||||||
| Function / homology |  Function and homology information Function and homology informationnebramycin 5' synthase / tobramycin biosynthetic process / kanamycin biosynthetic process / carboxyl- or carbamoyltransferase activity / ligase activity / iron ion binding / hydrolase activity / ATP binding Similarity search - Function | ||||||
| Biological species |  Streptoalloteichus tenebrarius (bacteria) Streptoalloteichus tenebrarius (bacteria) | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.2 Å MOLECULAR REPLACEMENT / Resolution: 2.2 Å | ||||||
 Authors Authors | Parthier, C. / Stubbs, M.T. / Goerlich, S. / Jaenecke, F. | ||||||
 Citation Citation |  Journal: Angew.Chem.Int.Ed.Engl. / Year: 2012 Journal: Angew.Chem.Int.Ed.Engl. / Year: 2012Title: The O-Carbamoyltransferase TobZ Catalyzes an Ancient Enzymatic Reaction. Authors: Parthier, C. / Gorlich, S. / Jaenecke, F. / Breithaupt, C. / Brauer, U. / Fandrich, U. / Clausnitzer, D. / Wehmeier, U.F. / Bottcher, C. / Scheel, D. / Stubbs, M.T. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  3vet.cif.gz 3vet.cif.gz | 242.8 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb3vet.ent.gz pdb3vet.ent.gz | 191.9 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  3vet.json.gz 3vet.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/ve/3vet https://data.pdbj.org/pub/pdb/validation_reports/ve/3vet ftp://data.pdbj.org/pub/pdb/validation_reports/ve/3vet ftp://data.pdbj.org/pub/pdb/validation_reports/ve/3vet | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  3venC  3veoSC  3verC  3vesC  3vewC  3vexC  3vezC  3vf2C  3vf4C  4e1bC C: citing same article ( S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
-Protein , 1 types, 1 molecules A
| #1: Protein | Mass: 63417.145 Da / Num. of mol.: 1 / Mutation: F35L Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Streptoalloteichus tenebrarius (bacteria) Streptoalloteichus tenebrarius (bacteria)Strain: DSM40770T / Gene: tacA, tobZ / Plasmid: pUWL201PW / Production host:  Streptomyces lividans (bacteria) / Strain (production host): TK24 Streptomyces lividans (bacteria) / Strain (production host): TK24References: UniProt: Q70IY1, Transferases; Transferring one-carbon groups; Carboxy- and carbamoyltransferases |
|---|
-Non-polymers , 8 types, 296 molecules 
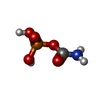













| #2: Chemical | | #3: Chemical | ChemComp-CP / | #4: Chemical | ChemComp-TOY / | #5: Chemical | ChemComp-MN / | #6: Chemical | ChemComp-K / | #7: Chemical | ChemComp-FE2 / | #8: Chemical | ChemComp-EDO / | #9: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.12 Å3/Da / Density % sol: 60.6 % |
|---|---|
| Crystal grow | Temperature: 289 K / Method: vapor diffusion, hanging drop / pH: 6.5 Details: 1.6 M sodium potassium tartrate, 0.1 M MES, pH 6.5, VAPOR DIFFUSION, HANGING DROP, temperature 289K |
-Data collection
| Diffraction | Mean temperature: 100 K | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  BESSY BESSY  / Beamline: 14.1 / Wavelength: 0.9184 Å / Beamline: 14.1 / Wavelength: 0.9184 Å | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Detector | Type: RAYONIX MX-225 / Detector: CCD / Date: Nov 25, 2009 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation | Monochromator: Double crystal Si(111) / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Radiation wavelength | Wavelength: 0.9184 Å / Relative weight: 1 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection | Resolution: 2.2→34.041 Å / Num. all: 42176 / Num. obs: 42176 / % possible obs: 99.961 % / Observed criterion σ(F): 0 / Observed criterion σ(I): -3 / Redundancy: 9.6 % / Biso Wilson estimate: 37.968 Å2 / Rmerge(I) obs: 0.075 / Net I/σ(I): 22.23 | |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Reflection shell |
|
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 3VEO Resolution: 2.2→34.041 Å / Occupancy max: 1 / Occupancy min: 0.38 / SU ML: 0.27 / Cross valid method: THROUGHOUT / σ(F): 1.56 / Phase error: 19.65 / Stereochemistry target values: ML
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Shrinkage radii: 0.95 Å / VDW probe radii: 1.2 Å / Solvent model: FLAT BULK SOLVENT MODEL / Bsol: 31.48 Å2 / ksol: 0.358 e/Å3 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso max: 124.23 Å2 / Biso mean: 35.7308 Å2 / Biso min: 4.57 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.2→34.041 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Refine-ID: X-RAY DIFFRACTION / Total num. of bins used: 28 / % reflection obs: 100 %
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS params. | Method: refined / Refine-ID: X-RAY DIFFRACTION
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement TLS group |
|
 Movie
Movie Controller
Controller












 PDBj
PDBj












