| 登録情報 | データベース: PDB / ID: 3sac
|
|---|
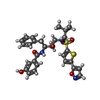
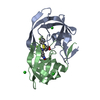
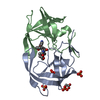
| タイトル | Crystal structure of wild-type HIV-1 protease in complex with AF80 |
|---|
 要素 要素 | Protease |
|---|
 キーワード キーワード | HYDROLASE/HYDROLASE INHIBITOR / HIV-1 protease / drug resistance / drug design / protease inhibitors / AIDS / aspartyl protease / HYDROLASE-HYDROLASE INHIBITOR complex |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
aspartic-type endopeptidase activity / proteolysis類似検索 - 分子機能 Retropepsin-like catalytic domain / Retropepsins / Retroviral aspartyl protease / Aspartyl protease, retroviral-type family profile. / Peptidase A2A, retrovirus, catalytic / Cathepsin D, subunit A; domain 1 / Acid Proteases / Aspartic peptidase, active site / Eukaryotic and viral aspartyl proteases active site. / Aspartic peptidase domain superfamily ...Retropepsin-like catalytic domain / Retropepsins / Retroviral aspartyl protease / Aspartyl protease, retroviral-type family profile. / Peptidase A2A, retrovirus, catalytic / Cathepsin D, subunit A; domain 1 / Acid Proteases / Aspartic peptidase, active site / Eukaryotic and viral aspartyl proteases active site. / Aspartic peptidase domain superfamily / Beta Barrel / Mainly Beta類似検索 - ドメイン・相同性 3-HYDROXY-N-{(2S,3R)-3-HYDROXY-4-[(2-METHYLPROPYL){[5-(1,2-OXAZOL-5-YL)THIOPHEN-2-YL]SULFONYL}AMINO]-1-PHENYLBUTAN-2-YL}BENZAMIDE / Chem-AF8 / PHOSPHATE ION / Protease類似検索 - 構成要素 |
|---|
| 生物種 |   Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.5 Å 分子置換 / 解像度: 1.5 Å |
|---|
 データ登録者 データ登録者 | Schiffer, C.A. / Nalam, M.N.L. |
|---|
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Published
タイトル: Protease Inhibitors that protrude out from substrate envelope are more susceptible to developing drug resistance
著者: Altman, M.D. / Nalam, M.N.L. / Ali, A. / Cao, H. / Rana, T.M. / Schiffer, C.A. / Tidor, B. |
|---|
| 履歴 | | 登録 | 2011年6月2日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2012年6月6日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2012年12月12日 | Group: Other |
|---|
| 改定 1.2 | 2017年11月8日 | Group: Refinement description / カテゴリ: software
Item: _software.classification / _software.contact_author ..._software.classification / _software.contact_author / _software.contact_author_email / _software.date / _software.language / _software.location / _software.name / _software.type / _software.version |
|---|
| 改定 1.3 | 2023年9月13日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.5 Å
分子置換 / 解像度: 1.5 Å  データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 3sac.cif.gz
3sac.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb3sac.ent.gz
pdb3sac.ent.gz PDB形式
PDB形式 3sac.json.gz
3sac.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 3sac_validation.pdf.gz
3sac_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 3sac_full_validation.pdf.gz
3sac_full_validation.pdf.gz 3sac_validation.xml.gz
3sac_validation.xml.gz 3sac_validation.cif.gz
3sac_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/sa/3sac
https://data.pdbj.org/pub/pdb/validation_reports/sa/3sac ftp://data.pdbj.org/pub/pdb/validation_reports/sa/3sac
ftp://data.pdbj.org/pub/pdb/validation_reports/sa/3sac リンク
リンク 集合体
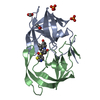
集合体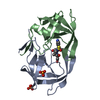
 要素
要素
 Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
Human immunodeficiency virus 1 (ヒト免疫不全ウイルス)
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X29A / 波長: 1.075
/ ビームライン: X29A / 波長: 1.075  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー













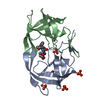
















 PDBj
PDBj