+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3j07 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Model of a 24mer alphaB-crystallin multimer | ||||||
 要素 要素 | Alpha-crystallin B chain | ||||||
 キーワード キーワード | CHAPERONE / sHSP | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報microtubule polymerization or depolymerization / negative regulation of intracellular transport / apoptotic process involved in morphogenesis / regulation of programmed cell death / cardiac myofibril / tubulin complex assembly / structural constituent of eye lens / negative regulation of amyloid fibril formation / M band / lens development in camera-type eye ...microtubule polymerization or depolymerization / negative regulation of intracellular transport / apoptotic process involved in morphogenesis / regulation of programmed cell death / cardiac myofibril / tubulin complex assembly / structural constituent of eye lens / negative regulation of amyloid fibril formation / M band / lens development in camera-type eye / muscle organ development / actin filament bundle / negative regulation of reactive oxygen species metabolic process / HSF1-dependent transactivation / negative regulation of protein-containing complex assembly / stress-activated MAPK cascade / muscle contraction / synaptic membrane / response to hydrogen peroxide / cellular response to gamma radiation / negative regulation of cell growth / Z disc / unfolded protein binding / protein folding / response to estradiol / amyloid-beta binding / response to heat / protein refolding / perikaryon / microtubule binding / dendritic spine / response to hypoxia / lysosome / protein stabilization / axon / negative regulation of gene expression / negative regulation of DNA-templated transcription / negative regulation of apoptotic process / protein-containing complex binding / structural molecule activity / cell surface / protein homodimerization activity / protein-containing complex / mitochondrion / extracellular exosome / nucleoplasm / metal ion binding / identical protein binding / nucleus / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 個体NMR /  溶液散乱 / 電子顕微鏡法 / 単粒子再構成法 / simulated annealing, molecular dynamics, torsion angle dynamics / ネガティブ染色法 / 解像度: 20 Å 溶液散乱 / 電子顕微鏡法 / 単粒子再構成法 / simulated annealing, molecular dynamics, torsion angle dynamics / ネガティブ染色法 / 解像度: 20 Å | ||||||
 データ登録者 データ登録者 | Jehle, S. / Vollmar, B. / Bardiaux, B. / Dove, K.K. / Rajagopal, P. / Gonen, T. / Oschkinat, H. / Klevit, R.E. | ||||||
 引用 引用 |  ジャーナル: Proc Natl Acad Sci U S A / 年: 2009 ジャーナル: Proc Natl Acad Sci U S A / 年: 2009タイトル: The eye lens chaperone alpha-crystallin forms defined globular assemblies. 著者: Jirka Peschek / Nathalie Braun / Titus M Franzmann / Yannis Georgalis / Martin Haslbeck / Sevil Weinkauf / Johannes Buchner /  要旨: Alpha-crystallins are molecular chaperones that protect vertebrate eye lens proteins from detrimental protein aggregation. alphaB-Crystallin, 1 of the 2 alpha-crystallin isoforms, is also associated ...Alpha-crystallins are molecular chaperones that protect vertebrate eye lens proteins from detrimental protein aggregation. alphaB-Crystallin, 1 of the 2 alpha-crystallin isoforms, is also associated with myopathies and neuropathological diseases. Despite the importance of alpha-crystallins in protein homeostasis, only little is known about their quaternary structures because of their seemingly polydisperse nature. Here, we analyzed the structures of recombinant alpha-crystallins using biophysical methods. In contrast to previous reports, we show that alphaB-crystallin assembles into defined oligomers consisting of 24 subunits. The 3-dimensional (3D) reconstruction of alphaB-crystallin by electron microscopy reveals a sphere-like structure with large openings to the interior of the protein. alphaA-Crystallin forms, in addition to complexes of 24 subunits, also smaller oligomers and large clusters consisting of individual oligomers. This propensity might explain the previously reported polydisperse nature of alpha-crystallin. #1:  ジャーナル: Nat Struct Mol Biol / 年: 2010 ジャーナル: Nat Struct Mol Biol / 年: 2010タイトル: Solid-state NMR and SAXS studies provide a structural basis for the activation of alphaB-crystallin oligomers. 著者: Stefan Jehle / Ponni Rajagopal / Benjamin Bardiaux / Stefan Markovic / Ronald Kühne / Joseph R Stout / Victoria A Higman / Rachel E Klevit / Barth-Jan van Rossum / Hartmut Oschkinat /  要旨: The small heat shock protein alphaB-crystallin (alphaB) contributes to cellular protection against stress. For decades, high-resolution structural studies on oligomeric alphaB have been confounded by ...The small heat shock protein alphaB-crystallin (alphaB) contributes to cellular protection against stress. For decades, high-resolution structural studies on oligomeric alphaB have been confounded by its polydisperse nature. Here, we present a structural basis of oligomer assembly and activation of the chaperone using solid-state NMR and small-angle X-ray scattering (SAXS). The basic building block is a curved dimer, with an angle of approximately 121 degrees between the planes of the beta-sandwich formed by alpha-crystallin domains. The highly conserved IXI motif covers a substrate binding site at pH 7.5. We observe a pH-dependent modulation of the interaction of the IXI motif with beta4 and beta8, consistent with a pH-dependent regulation of the chaperone function. N-terminal region residues Ser59-Trp60-Phe61 are involved in intermolecular interaction with beta3. Intermolecular restraints from NMR and volumetric restraints from SAXS were combined to calculate a model of a 24-subunit alphaB oligomer with tetrahedral symmetry. | ||||||
| 履歴 |
| ||||||
| Remark 0 | THIS ENTRY 3J07 CONTAINS A STRUCTURAL MODEL FIT TO AN ELECTRON MICROSCOPY MAP (EMD-1776) DETERMINED ...THIS ENTRY 3J07 CONTAINS A STRUCTURAL MODEL FIT TO AN ELECTRON MICROSCOPY MAP (EMD-1776) DETERMINED ORIGINALLY BY AUTHORS: J.PESCHEK, N.BRAUN, T.M.FRANZMANN, Y.GEORGALIS, M.HASLBECK, S.WEINKAUF, J.BUCHNER |
- 構造の表示
構造の表示
| ムービー |
 ムービービューア ムービービューア |
|---|---|
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3j07.cif.gz 3j07.cif.gz | 872.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3j07.ent.gz pdb3j07.ent.gz | 719 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3j07.json.gz 3j07.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3j07_validation.pdf.gz 3j07_validation.pdf.gz | 500.4 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3j07_full_validation.pdf.gz 3j07_full_validation.pdf.gz | 738.4 KB | 表示 | |
| XML形式データ |  3j07_validation.xml.gz 3j07_validation.xml.gz | 113 KB | 表示 | |
| CIF形式データ |  3j07_validation.cif.gz 3j07_validation.cif.gz | 154.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/j0/3j07 https://data.pdbj.org/pub/pdb/validation_reports/j0/3j07 ftp://data.pdbj.org/pub/pdb/validation_reports/j0/3j07 ftp://data.pdbj.org/pub/pdb/validation_reports/j0/3j07 | HTTPS FTP |
-関連構造データ
| 関連構造データ | 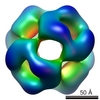 1776M M: このデータのモデリングに利用したマップデータ |
|---|---|
| 類似構造データ | |
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 20191.930 Da / 分子数: 24 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: CRYAB, CRYA2 / プラスミド: pET16b / 発現宿主: Homo sapiens (ヒト) / 遺伝子: CRYAB, CRYA2 / プラスミド: pET16b / 発現宿主:  |
|---|
-実験情報
-実験
| 実験 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| EM実験 | 試料の集合状態: PARTICLE / 3次元再構成法: 単粒子再構成法 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| NMR実験 |
|
- 試料調製
試料調製
| 構成要素 | 名称: 24mer alphaB-crystallin multimer / タイプ: COMPLEX / 詳細: polydisperse oligomer | ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 緩衝液 | pH: 7.4 / 詳細: 137mM NaCl, 2.7mM KCl, 12 mM PBS | ||||||||||||||||||||||||||||||||||||
| 試料 | 包埋: NO / シャドウイング: NO / 染色: YES / 凍結: NO | ||||||||||||||||||||||||||||||||||||
| 染色 | タイプ: NEGATIVE / 染色剤: Uranyl Acetate | ||||||||||||||||||||||||||||||||||||
| 結晶 | 解説: HOMOLOGY MODELS AND SPARSE SOLID-STATE NMR DISTANCE RESTRAINTS WERE USED TO DEFINE THE SECONDARY STRUCTURE OF THE N-TERMINAL DOMAIN (UNP RESIDUES 1-65). THE RESTRAINTS ARE GIVEN IN THE ...解説: HOMOLOGY MODELS AND SPARSE SOLID-STATE NMR DISTANCE RESTRAINTS WERE USED TO DEFINE THE SECONDARY STRUCTURE OF THE N-TERMINAL DOMAIN (UNP RESIDUES 1-65). THE RESTRAINTS ARE GIVEN IN THE SUPPORTING INFORMATION OF THE PRIMARY CITATION. A 2D 1H-13C INEPT SPECTRUM WAS RECORDED AND ANALYZED IN ORDER TO IDENTIFY FLEXIBLE RESIDUES IN THE EXTREME N- AND C-TERMINI. SEVERAL MODELS WERE PREPARED WITH DIFFERENT POSITIONS OF FLEXIBLE RESIDUES IN THE INSIDE OF THE SPHERICAL MULTIMER TO FIT THE RADIUS OF GYRATION (RG) MEASURED BY SAXS. THE MODEL THAT BEST FIT THE RG WAS CHOSEN FOR DEPOSITION. | ||||||||||||||||||||||||||||||||||||
| 詳細 |
| ||||||||||||||||||||||||||||||||||||
| 試料 |
| ||||||||||||||||||||||||||||||||||||
| 試料状態 | pH: 7.6 / 温度: 270 K |
-データ収集
| 顕微鏡 | モデル: JEOL 100CX / 日付: 2008年2月6日 | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 電子銃 | 電子線源: OTHER / 加速電圧: 100 kV / 照射モード: FLOOD BEAM | |||||||||||||||||||||||||
| 電子レンズ | モード: BRIGHT FIELD / 倍率(公称値): 50000 X / 最大 デフォーカス(公称値): 1200 nm / 最小 デフォーカス(公称値): 900 nm | |||||||||||||||||||||||||
| 撮影 | フィルム・検出器のモデル: KODAK SO-163 FILM | |||||||||||||||||||||||||
| NMRスペクトロメーター |
| |||||||||||||||||||||||||
| Soln scatter | タイプ: x-ray / Buffer name: phosphate, 100 mM NaCl / Conc. range: 0.1-12.6 mg/mL / Data reduction software list: CBF-PRIMUS / 検出器タイプ: 2D MAR345 / Mean guiner radius: 5.6 nm / Mean guiner radius esd: 0.2 nm / Num. of time frames: 1 / Protein length: 14.5 / Sample pH: 7.5 / Source beamline: X33 / Source class: Y / Source type: DESY HAMBURG X33 BEAMLINE / 温度: 293 K |
- 解析
解析
| 対称性 | 点対称性: T (正4面体型対称) | ||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 3次元再構成 | 解像度: 20 Å / 解像度の算出法: FSC 0.5 CUT-OFF / 粒子像の数: 2565 / 対称性のタイプ: POINT | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子モデル構築 | PDB-ID: 2KLR Accession code: 2KLR / Source name: PDB / タイプ: experimental model | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST
| ||||||||||||||||||||||||||||||||||||||||||||||||||||
| NMR software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | 手法: simulated annealing, molecular dynamics, torsion angle dynamics ソフトェア番号: 11 詳細: THREE DIMERS CONNECTED BY THEIR C-TERMINAL IXI MOTIF WERE FITTED BACK-TO-BACK IN THE EM DENSITY USING CHIMERA. A HEXAMER WAS BUILT FROM MODEL #7 OF PDB ENTRY 2KLR USING NONCRYSTALLOGRAPHIC ...詳細: THREE DIMERS CONNECTED BY THEIR C-TERMINAL IXI MOTIF WERE FITTED BACK-TO-BACK IN THE EM DENSITY USING CHIMERA. A HEXAMER WAS BUILT FROM MODEL #7 OF PDB ENTRY 2KLR USING NONCRYSTALLOGRAPHIC SYMMETRY RESTRAINTS GIVEN IN THE PDB FILE. DIMERS WERE CONNECTED BY THEIR C-TERMINAL DOMAIN CONTAINING THE IXI-MOTIF, AND THE LINKER (UNP RESIDUES 151-155) WAS ENERGY MINIMIZED USING XPLOR-NIH. THE BETA1/BETA2(FREE) STRUCTURAL SEGMENT WAS PLACED INTO THE EM DENSITY TO FULFILL THE CROSSLINKS FOR A57, S59, AND T63, SIMILAR TO PDB ENTRY 1VLQ. ALPHA2 HELICES OF TWO MONOMERS WERE DOCKED, DEFINING RESIDUES L32, L33, L37, AND F38 AS INTERACTING RESIDUES USING HADDOCK. THE ALPHA2 HELICAL COMPLEX WAS PLACED IN WEAK EM DENSITY AT THE THREE-FOLD AXIS. THE FLEXIBLE C- AND N-TERMINAL RESIDUES, INCLUDING PARTS OF HELIX ALPHA1, WERE PLACED INSIDE THE 24MER TO FIT THE RADIUS OF GYRATION, MEASURED BY SMALL-ANGLE X-RAY SCATTERING IN AN ITERATIVE PROCESS. ALL FRAGMENTS WERE CONNECTED USING DISCOVERY STUDIO 1.6 (ACCELRYS) AND THE LINKERS WERE ENERGY MINIMIZED. BACK PROJECTIONS WERE CALCULATED FOR MODELS OF ALPHAB MULTIMERS CONTAINING 24, 26, AND 28 SUBUNITS USING THE PROGRAM SPIDER. THE CALCULATED PROJECTIONS ARE SIMILAR TO EACH OTHER, TO THE CLASS SUM IMAGES FROM COLLECTED EM DATA, AND TO THOSE CALCULATED FROM THE PUBLISHED EM DENSITY (EMD-1776). DETAILS ARE GIVEN IN THE PRIMARY CITATION. | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest energy | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 200 / 登録したコンフォーマーの数: 1 | ||||||||||||||||||||||||||||||||||||||||||||||||||||
| Soln scatter model | 手法: FITTING OF MODELS WITH SAXS DATA; コンフォーマー選択の基準: BEST FIT WITH NMR, SAXS, AND EM DATA 詳細: TO DETERMINE HETEROGENEITY, A PYTHON SCRIPT WAS USED TO MODULATE AND FIT THE THEORETICAL SAXS CURVE OF THE MODEL WITH THE EXPERIMENTAL DATA. THE ALGORITHM THAT WAS APPLIED HAS BEEN PUBLISHED ...詳細: TO DETERMINE HETEROGENEITY, A PYTHON SCRIPT WAS USED TO MODULATE AND FIT THE THEORETICAL SAXS CURVE OF THE MODEL WITH THE EXPERIMENTAL DATA. THE ALGORITHM THAT WAS APPLIED HAS BEEN PUBLISHED BY MITTELBACH, R. AND GLATTER, O. (1998) IN J. APPL. CRYSTALLOGR. 31:600-608. FOR DETAILS SEE PRIMARY CITATION. Num. of conformers calculated: 10 / Num. of conformers submitted: 1 / 代表コンフォーマー: 1 Software author list: PETOUKHOV, M.V.; SVERGUN, D.I.; SCHWIETERS, C.D. PETTERSON, E.F.; GODDARD, T.D.; ACCELRYS Software list: CHIMERA, PRIMUS, GNOM, XPLOR-NIH, CRYSOL, DISCOVERY STUDIO 1.3; |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj


