+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 3fx8 | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | Distinct recognition of three-way DNA junctions by a thioester variant of a metallo-supramolecular cylinder ('helicate') | ||||||||||||||||||
 要素 要素 | (5'-D(* キーワード キーワードDNA / Self-assembly / DNA-based nanomaterial | 機能・相同性 | Chem-5PM / : / DNA |  機能・相同性情報 機能・相同性情報手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  多波長異常分散 / 解像度: 2.44 Å 多波長異常分散 / 解像度: 2.44 Å  データ登録者 データ登録者Boer, D.R. / Uson, I. / Coll, M. |  引用 引用 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2010 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2010タイトル: Self-Assembly of Functionalizable Two-Component 3D DNA Arrays through the Induced Formation of DNA Three-Way-Junction Branch Points by Supramolecular Cylinders. 著者: Boer, D.R. / Kerckhoffs, J.M. / Parajo, Y. / Pascu, M. / Uson, I. / Lincoln, P. / Hannon, M.J. / Coll, M. #1:  ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2006 ジャーナル: Angew.Chem.Int.Ed.Engl. / 年: 2006タイトル: Molecular recognition of a three-way DNA junction by a metallosupramolecular helicate. 著者: Oleksy, A. / Blanco, A.G. / Boer, R. / Uson, I. / Aymami, J. / Rodger, A. / Hannon, M.J. / Coll, M. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  3fx8.cif.gz 3fx8.cif.gz | 15.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb3fx8.ent.gz pdb3fx8.ent.gz | 9.2 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  3fx8.json.gz 3fx8.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  3fx8_validation.pdf.gz 3fx8_validation.pdf.gz | 762.1 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  3fx8_full_validation.pdf.gz 3fx8_full_validation.pdf.gz | 763.8 KB | 表示 | |
| XML形式データ |  3fx8_validation.xml.gz 3fx8_validation.xml.gz | 3.1 KB | 表示 | |
| CIF形式データ |  3fx8_validation.cif.gz 3fx8_validation.cif.gz | 3.4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/fx/3fx8 https://data.pdbj.org/pub/pdb/validation_reports/fx/3fx8 ftp://data.pdbj.org/pub/pdb/validation_reports/fx/3fx8 ftp://data.pdbj.org/pub/pdb/validation_reports/fx/3fx8 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 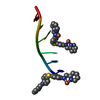
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| |||||||||||||||
| 単位格子 |
| |||||||||||||||
| Components on special symmetry positions |
| |||||||||||||||
| 詳細 | A THREE-WAY JUNCTION (CHAINS A AND C) WITH DISCONTINUOUS DNA STRANDS CAN BE GENERATED THROUGH THE FOLLOWING TRANSFORMATIONS ON CHAIN C: [Z+1/2,-X+1/2,-Y+(0 1 1)], [Y+1/2,-Z+1/2,-X+(-1 1 1)], [-X, Y+1/2,-Z+1/2+(0 1 0)], [-Z,X+1/2,-Y+1/2+(0 1 1)], [Y,Z,X+(0 2 1)] AND THE FOLLOWING TRANSFORMATIONS ON CHAIN A: [-Z,X+1/2,-Y+1/2 +(0 1 1)], [Y+1/2,-Z+1/2,-X +(-1 1 1)] |
- 要素
要素
| #1: DNA鎖 | ( 分子量: 1809.217 Da / 分子数: 1 / 由来タイプ: 合成 詳細: Synthetic hexanucleotide. The sequence is palindromic | ||||
|---|---|---|---|---|---|
| #2: 化合物 | ChemComp-FE2 / #3: 化合物 | 非ポリマーの詳細 | LIGAND 5PM C 111 WITH TWO IRON ATOMS FE2 C 101 AND C 102 IS THE M HELICAL ENANTIOMER OF [FE2L3], ...LIGAND 5PM C 111 WITH TWO IRON ATOMS FE2 C 101 AND C 102 IS THE M HELICAL ENANTIOMER | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 5.5 Å3/Da / 溶媒含有率: 77.64 % | ||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 8.5 詳細: 1 microliter of 10 mM cylinder, 1 microliter of 3 mM DNA, 2 microliters of crystallization buffer (10 mM Magnesium chloride, 5% v/v Isopropanol, 50 mM Tris-HCl), pH 8.5, VAPOR DIFFUSION, ...詳細: 1 microliter of 10 mM cylinder, 1 microliter of 3 mM DNA, 2 microliters of crystallization buffer (10 mM Magnesium chloride, 5% v/v Isopropanol, 50 mM Tris-HCl), pH 8.5, VAPOR DIFFUSION, SITTING DROP, temperature 293K | ||||||||||||||||||||||||||||
| 溶液の組成 |
|
-データ収集
| 回折 | 平均測定温度: 100 K | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ESRF ESRF  / ビームライン: ID29 / 波長: 1.7366, 1.7410, 1.7311 / ビームライン: ID29 / 波長: 1.7366, 1.7410, 1.7311 | ||||||||||||
| 検出器 | タイプ: ADSC QUANTUM 315 / 検出器: CCD / 日付: 2006年2月23日 | ||||||||||||
| 放射 | モノクロメーター: Si(111) / プロトコル: MAD / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray | ||||||||||||
| 放射波長 |
| ||||||||||||
| 反射 | 解像度: 2.44→24.63 Å / Num. all: 1573 / Num. obs: 1573 / % possible obs: 99.4 % / Observed criterion σ(F): 0 / Observed criterion σ(I): -3 / 冗長度: 8.4 % / Biso Wilson estimate: 94 Å2 / Rmerge(I) obs: 0.041 / Net I/σ(I): 36.1 | ||||||||||||
| 反射 シェル | 解像度: 2.44→2.58 Å / 冗長度: 5.1 % / Rmerge(I) obs: 0.447 / Mean I/σ(I) obs: 2.2 / Num. unique all: 228 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  多波長異常分散 / 解像度: 2.44→24.63 Å / Isotropic thermal model: isotropic 多波長異常分散 / 解像度: 2.44→24.63 Å / Isotropic thermal model: isotropic
| |||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: BABINET (SHELXL SWAT) | |||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 79.4 Å2 | |||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.44→24.63 Å
| |||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 2.44→2.55 Å /
|
 ムービー
ムービー コントローラー
コントローラー














 PDBj
PDBj