+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 376d | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| タイトル | A ZIPPER-LIKE DNA DUPLEX D(GCGAAAGCT) | ||||||||||||||||||
 要素 要素 | DNA (5'-D(* キーワード キーワードDNA / RIGHT HANDED DNA / DOUBLE HELIX / OVERHANGING BASE / MODIFIED / MISMATCHED | 機能・相同性 | COBALT HEXAMMINE(III) / DNA |  機能・相同性情報 機能・相同性情報手法 |  X線回折 / X線回折 /  シンクロトロン / MAD AT BROMINE EDGE / 解像度: 2.1 Å シンクロトロン / MAD AT BROMINE EDGE / 解像度: 2.1 Å  データ登録者 データ登録者Cruse, W.B.T. / Shepard, W. / Prange, T. / delalFortelle, E. / Fourme, R. |  引用 引用 ジャーナル: Structure / 年: 1998 ジャーナル: Structure / 年: 1998タイトル: A zipper-like duplex in DNA: the crystal structure of d(GCGAAAGCT) at 2.1 A resolution. 著者: Shepard, W. / Cruse, W.B. / Fourme, R. / de la Fortelle, E. / Prange, T. #1:  ジャーナル: Science / 年: 1997 ジャーナル: Science / 年: 1997タイトル: RNA Tertiary Structure Mediation by Adenosine Platforms 著者: Cate, J.H. / Gooding, A.R. / Podell, E. / Zhou, K. / Golden, B.L. / Szewczak, A.A. / Kundrot, C.E. / Cech, T.R. / Doudna, J.A. #2:  ジャーナル: Structure / 年: 1997 ジャーナル: Structure / 年: 1997タイトル: Solution Structure of a Metal-binding Site in the Major Groove of RNA Complexed with Cobalt(III) Hexammine 著者: Kieft, J.S. / Tinoco Jr., I. #3:  ジャーナル: Nucleic Acids Res. / 年: 1989 ジャーナル: Nucleic Acids Res. / 年: 1989タイトル: Extraordinary Stable Structure of Short Single-Stranded DNA Fragments Containing a Specific Base Sequence: d(GCGAAAGC) 著者: Hirao, I. / Nichimura, Y. / Naraoka, T. / Watanabe, K. / Arata, Y. / Miura, K. #4:  ジャーナル: Biochemistry / 年: 1985 ジャーナル: Biochemistry / 年: 1985タイトル: Structural Basis for Stabilisation of Z-DNA by Cobalt Haxaammine and Magnesium Cations 著者: Geissner, R.V. / Quigley, G.J. / Wang, H.J. / van der Marel, A. / van Boom, J.H. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  376d.cif.gz 376d.cif.gz | 15.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb376d.ent.gz pdb376d.ent.gz | 9.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  376d.json.gz 376d.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  376d_validation.pdf.gz 376d_validation.pdf.gz | 329.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  376d_full_validation.pdf.gz 376d_full_validation.pdf.gz | 331.3 KB | 表示 | |
| XML形式データ |  376d_validation.xml.gz 376d_validation.xml.gz | 1.8 KB | 表示 | |
| CIF形式データ |  376d_validation.cif.gz 376d_validation.cif.gz | 2.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/76/376d https://data.pdbj.org/pub/pdb/validation_reports/76/376d ftp://data.pdbj.org/pub/pdb/validation_reports/76/376d ftp://data.pdbj.org/pub/pdb/validation_reports/76/376d | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 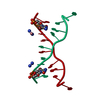
| ||||||||||
| 単位格子 |
| ||||||||||
| Components on special symmetry positions |
|
- 要素
要素
| #1: DNA鎖 | 分子量: 2843.733 Da / 分子数: 1 / 由来タイプ: 組換発現 |
|---|---|
| #2: 化合物 | ChemComp-NCO / |
| #3: 水 | ChemComp-HOH / |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 3 X線回折 / 使用した結晶の数: 3 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.3 Å3/Da / 溶媒含有率: 36 % | |||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 溶液の組成 |
| |||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶 | *PLUS 溶媒含有率: 36 % | |||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 4 ℃ / 手法: 蒸気拡散法 | |||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 276 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: LURE シンクロトロン / サイト: LURE  / ビームライン: DW21B / ビームライン: DW21B |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1996年9月1日 / 詳細: DOUBLE MONO + MULTILAYER FOCUS |
| 放射 | モノクロメーター: SI(III) / プロトコル: MAD / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 相対比: 1 |
| 反射 | 解像度: 2.1→15 Å / Num. obs: 1832 / % possible obs: 95 % / Observed criterion σ(I): 3 / Rmerge(I) obs: 0.053 / Net I/σ(I): 11.3 |
| 反射 シェル | 最高解像度: 2.1 Å / Rmerge(I) obs: 0.148 / Mean I/σ(I) obs: 4.2 / % possible all: 61 |
| 反射 | *PLUS 最高解像度: 2.1 Å / 最低解像度: 15 Å / % possible obs: 95 % / Observed criterion σ(I): 3 / 冗長度: 11.6 % / Num. measured all: 21345 |
| 反射 シェル | *PLUS 最高解像度: 2.1 Å / 最低解像度: 2.2 Å / Mean I/σ(I) obs: 4.2 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: MAD AT BROMINE EDGE 開始モデル: MANUALLY BUILT IN THE EXPERIMENTAL DENSITY 解像度: 2.1→15 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze | Luzzati coordinate error obs: 0.15 Å / Luzzati d res low obs: 15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.1→15 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称:  X-PLOR / 分類: refinement X-PLOR / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化 | *PLUS 最高解像度: 2.1 Å / 最低解像度: 15 Å | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS タイプ: x_angle_d |
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj