+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2y95 | ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
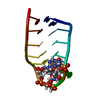
| タイトル | Solution structure of AUCG tetraloop hairpin found in human Xist RNA A-repeats essential for X-inactivation | ||||||||||||||||||||
 要素 要素 | 5'-R(* キーワード キーワードRNA / X CHROMOSOME INACTIVATION / RNA STEMLOOP | 機能・相同性 | : / RNA / RNA (> 10) |  機能・相同性情報 機能・相同性情報生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト)手法 | 溶液NMR / ARIA |  データ登録者 データ登録者Duszczyk, M.M. / Sattler, M. |  引用 引用 ジャーナル: RNA / 年: 2011 ジャーナル: RNA / 年: 2011タイトル: The Xist RNA A-Repeat Comprises a Novel Aucg Tetraloop Fold and a Platform for Multimerization. 著者: Duszczyk, M.M. / Wutz, A. / Rybin, V. / Sattler, M. #1: ジャーナル: Biomol.NMR Assign. / 年: 2012 タイトル: (1)H, (13)C, (15)N and (31)P Chemical Shift Assignments of a Human Xist RNA A-Repeat Tetraloop Hairpin Essential for X-Chromosome Inactivation. 著者: Duszczyk, M.M. / Sattler, M. 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2y95.cif.gz 2y95.cif.gz | 92.3 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2y95.ent.gz pdb2y95.ent.gz | 74.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2y95.json.gz 2y95.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2y95_validation.pdf.gz 2y95_validation.pdf.gz | 402.8 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2y95_full_validation.pdf.gz 2y95_full_validation.pdf.gz | 451.2 KB | 表示 | |
| XML形式データ |  2y95_validation.xml.gz 2y95_validation.xml.gz | 7.4 KB | 表示 | |
| CIF形式データ |  2y95_validation.cif.gz 2y95_validation.cif.gz | 8.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/y9/2y95 https://data.pdbj.org/pub/pdb/validation_reports/y9/2y95 ftp://data.pdbj.org/pub/pdb/validation_reports/y9/2y95 ftp://data.pdbj.org/pub/pdb/validation_reports/y9/2y95 | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 4492.739 Da / 分子数: 1 / 断片: HAIRPIN 1, RESIDUES 609-622 / 変異: YES / 由来タイプ: 合成 詳細: HAIRPIN 1 FROM THE 6TH HUMAN A-REPEAT CORRESPONDING TO NUCLEOTIDES 609-622 IN M97168.1 GENBANK ENTRY. THE THIRD G-C BASE-PAIR IS REPLACED BY C-G TO FACILITATE CHEMICAL SHIFT ASSIGNMENTS AND ...詳細: HAIRPIN 1 FROM THE 6TH HUMAN A-REPEAT CORRESPONDING TO NUCLEOTIDES 609-622 IN M97168.1 GENBANK ENTRY. THE THIRD G-C BASE-PAIR IS REPLACED BY C-G TO FACILITATE CHEMICAL SHIFT ASSIGNMENTS AND THE CLOSING G-U BASE PAIR IS REPLACED BY G-C 由来: (合成)  HOMO SAPIENS (ヒト) / 参照: GenBank: 340393 HOMO SAPIENS (ヒト) / 参照: GenBank: 340393 |
|---|---|
| 構成要素の詳細 | ENGINEERED RESIDUE IN CHAIN A, C 612 TO G ENGINEERED RESIDUE IN CHAIN A, G 619 TO C ENGINEERED ...ENGINEERED |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| NMR実験の詳細 | Text: 100 INITIAL STRUCTURES WERE CALCULATED USING ARIA, 20 WERE SELECTED FOR REFINEMENT WITH AMBER, OF WHICH 10 WERE SELECTED FOR DEPOSITION BASED ON LOW ENERGY CRITERIA AND AGREEMENT BETWEEN ...Text: 100 INITIAL STRUCTURES WERE CALCULATED USING ARIA, 20 WERE SELECTED FOR REFINEMENT WITH AMBER, OF WHICH 10 WERE SELECTED FOR DEPOSITION BASED ON LOW ENERGY CRITERIA AND AGREEMENT BETWEEN CALCULATED AND EXPERIMENTAL CHEMICAL SHIFTS |
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
|
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: ARIA / ソフトェア番号: 1 詳細: 20 ARIA-CALCULATED STRUCTURES WERE CHOSEN BASED UPON LOW RESTRAINT VIOLATION AND LOW ENERGY FOR REFINEMENT WITH AMBER | ||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATION, LOW ENERGY, AGREEMENT WITH CHEMICAL SHIFTS CALCULATED BY NUCHEMICS 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 10 |
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj





























 HSQC
HSQC