[English] 日本語
 Yorodumi
Yorodumi- PDB-2wlb: Adrenodoxin-like ferredoxin Etp1fd(516-618) of Schizosaccharomyce... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 2wlb | ||||||
|---|---|---|---|---|---|---|---|
| Title | Adrenodoxin-like ferredoxin Etp1fd(516-618) of Schizosaccharomyces pombe mitochondria | ||||||
 Components Components | ELECTRON TRANSFER PROTEIN 1, MITOCHONDRIAL | ||||||
 Keywords Keywords | ELECTRON TRANSPORT / IRON-SULFUR / MITOCHONDRIA / IRON / TRANSPORT / FERREDOXIN / ADRENODOXIN-LIKE / METAL-BINDING | ||||||
| Function / homology |  Function and homology information Function and homology informationHeme biosynthesis / heme a synthase / oxidoreductase activity, acting on NAD(P)H, heme protein as acceptor / heme A biosynthetic process / P450-containing electron transport chain / NADPH-hemoprotein reductase activity / mitochondrial [2Fe-2S] assembly complex / [2Fe-2S] cluster assembly / oxidoreductase activity, acting on NAD(P)H / iron-sulfur cluster binding ...Heme biosynthesis / heme a synthase / oxidoreductase activity, acting on NAD(P)H, heme protein as acceptor / heme A biosynthetic process / P450-containing electron transport chain / NADPH-hemoprotein reductase activity / mitochondrial [2Fe-2S] assembly complex / [2Fe-2S] cluster assembly / oxidoreductase activity, acting on NAD(P)H / iron-sulfur cluster binding / 2 iron, 2 sulfur cluster binding / mitochondrial inner membrane / mitochondrial matrix / mitochondrion / metal ion binding Similarity search - Function | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  MOLECULAR REPLACEMENT / Resolution: 2.6 Å MOLECULAR REPLACEMENT / Resolution: 2.6 Å | ||||||
 Authors Authors | Mueller, J.J. / Hannemann, F. / Schiffler, B. / Bernhardt, R. / Heinemann, U. | ||||||
 Citation Citation |  Journal: J.Inorg.Biochem. / Year: 2011 Journal: J.Inorg.Biochem. / Year: 2011Title: Structural and Thermodynamic Characterization of the Adrenodoxin-Like Domain of the Electron-Transfer Protein Etp1 from Schizosaccharomyces Pombe. Authors: Mueller, J.J. / Hannemann, F. / Schiffler, B. / Ewen, K.M. / Kappl, R. / Heinemann, U. / Bernhardt, R. | ||||||
| History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  2wlb.cif.gz 2wlb.cif.gz | 52.7 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb2wlb.ent.gz pdb2wlb.ent.gz | 37.6 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  2wlb.json.gz 2wlb.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/wl/2wlb https://data.pdbj.org/pub/pdb/validation_reports/wl/2wlb ftp://data.pdbj.org/pub/pdb/validation_reports/wl/2wlb ftp://data.pdbj.org/pub/pdb/validation_reports/wl/2wlb | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 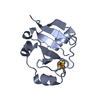
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 2 | 
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Unit cell |
| |||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Noncrystallographic symmetry (NCS) | NCS domain:
NCS domain segments:
NCS oper:
|
- Components
Components
| #1: Protein | Mass: 11306.626 Da / Num. of mol.: 2 / Fragment: ADRENODOXIN-LIKE DOMAIN, RESIDUES 516-618 Source method: isolated from a genetically manipulated source Source: (gene. exp.)  Production host:  #2: Chemical | #3: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 2 X-RAY DIFFRACTION / Number of used crystals: 2 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.83 Å3/Da / Density % sol: 56.6 % / Description: NONE |
|---|---|
| Crystal grow | Temperature: 277 K / Method: vapor diffusion, sitting drop / pH: 7.4 Details: VAPOUR DIFFUSION, SITTING DROP, 4 DEGR. CENTIGRADE. RESERVOIR: 0.04M HEPES,1.8M NA-CITRATE, 3% GLYCEROL,PH7.0. PROTEIN SOLUTION: 18.4MG/ML PROTEIN, 20MM POTASSIUMPHOSPHATE,PH7.4. 0.3 PLUS 0.3 MICROLITER DROPLET. |
-Data collection
| Diffraction | Mean temperature: 278 K |
|---|---|
| Diffraction source | Source:  ROTATING ANODE / Type: RIGAKU RU H2B / Wavelength: 1.5418 ROTATING ANODE / Type: RIGAKU RU H2B / Wavelength: 1.5418 |
| Detector | Type: MAR scanner 345 mm plate / Detector: IMAGE PLATE / Date: Apr 6, 2004 |
| Radiation | Monochromator: OSMIC MIRROR / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1.5418 Å / Relative weight: 1 |
| Reflection | Resolution: 2.6→20 Å / Num. obs: 8006 / % possible obs: 99.5 % / Redundancy: 3.4 % / Biso Wilson estimate: 56.1 Å2 / Rmerge(I) obs: 0.11 / Net I/σ(I): 8.5 |
| Reflection shell | Resolution: 2.6→2.78 Å / Redundancy: 3.5 % / Rmerge(I) obs: 0.51 / Mean I/σ(I) obs: 3.4 / % possible all: 92.4 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRIES 1E6E,1AYF,1L6V,2BT6 Resolution: 2.6→20 Å / Cor.coef. Fo:Fc: 0.962 / Cor.coef. Fo:Fc free: 0.935 / SU B: 10.626 / SU ML: 0.203 / Cross valid method: THROUGHOUT / ESU R: 0.463 / ESU R Free: 0.276 / Stereochemistry target values: MAXIMUM LIKELIHOOD / Details: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | Ion probe radii: 0.8 Å / Shrinkage radii: 0.8 Å / VDW probe radii: 1.2 Å / Solvent model: BABINET MODEL WITH MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 45.15 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.6→20 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
|
 Movie
Movie Controller
Controller














 PDBj
PDBj










