| 登録情報 | データベース: PDB / ID: 2w85
|
|---|
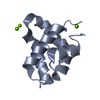
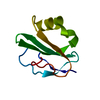
| タイトル | Structure of Pex14 in complex with Pex19 |
|---|
 要素 要素 | - PEROXIN-19
- PEROXISOMAL MEMBRANE ANCHOR PROTEIN PEX14
|
|---|
 キーワード キーワード | PROTEIN TRANSPORT / ZELLWEGER SYNDROME / ALTERNATIVE SPLICING / TRANSLOCATION / PHOSPHOPROTEIN / PROTEIN COMPLEX / PEROXISOME BIOGENESIS DISORDER / PEROXISOME TARGETING SIGNAL / PEROXISOME / PRENYLATION / LIPOPROTEIN / POLYMORPHISM / PTS / MEMBRANE / PEROXISOME IMPORT / PEROXISOME BIOGENESIS / RECEPTOR-CARGO COMPLEX |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
peroxisome membrane biogenesis / peroxisome membrane class-1 targeting sequence binding / peroxisome transport along microtubule / establishment of protein localization to peroxisome / negative regulation of lipid binding / protein import into peroxisome matrix, substrate release / protein import into peroxisome matrix, translocation / peroxisome membrane targeting sequence binding / protein import into peroxisome membrane / protein targeting to peroxisome ...peroxisome membrane biogenesis / peroxisome membrane class-1 targeting sequence binding / peroxisome transport along microtubule / establishment of protein localization to peroxisome / negative regulation of lipid binding / protein import into peroxisome matrix, substrate release / protein import into peroxisome matrix, translocation / peroxisome membrane targeting sequence binding / protein import into peroxisome membrane / protein targeting to peroxisome / peroxisomal importomer complex / protein import into peroxisome matrix / microtubule anchoring / protein import into peroxisome matrix, docking / Class I peroxisomal membrane protein import / peroxisome organization / protein carrier chaperone / ABC transporters in lipid homeostasis / peroxisome fission / peroxisomal membrane / protein transmembrane transporter activity / beta-tubulin binding / negative regulation of DNA-binding transcription factor activity / negative regulation of protein binding / : / Peroxisomal protein import / cellular response to reactive oxygen species / brush border membrane / fibrillar center / transcription corepressor activity / E3 ubiquitin ligases ubiquitinate target proteins / peroxisome / ATPase binding / protein-containing complex assembly / protein-macromolecule adaptor activity / microtubule binding / protein stabilization / signaling receptor binding / negative regulation of DNA-templated transcription / protein-containing complex / nucleoplasm / identical protein binding / nucleus / membrane / cytosol / cytoplasm類似検索 - 分子機能 Pex19 protein / Pex19, C-terminal domain superfamily / Pex19 protein family / Peroxisome membrane anchor protein Pex14p, N-terminal / Peroxisomal membrane protein 14 / Pex14 N-terminal domain / Winged helix-like DNA-binding domain superfamily/Winged helix DNA-binding domain / Arc Repressor Mutant, subunit A / Winged helix-like DNA-binding domain superfamily / Orthogonal Bundle / Mainly Alpha類似検索 - ドメイン・相同性 Peroxisomal membrane protein PEX14 / Peroxisomal biogenesis factor 19類似検索 - 構成要素 |
|---|
| 生物種 |  HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) |
|---|
| 手法 | 溶液NMR / torsion angle dynamics |
|---|
 データ登録者 データ登録者 | Neufeld, C. / Filipp, F.V. / Simon, B. / Neuhaus, A. / Schueller, N. / David, C. / Kooshapur, H. / Madl, T. / Erdmann, R. / Schliebs, W. ...Neufeld, C. / Filipp, F.V. / Simon, B. / Neuhaus, A. / Schueller, N. / David, C. / Kooshapur, H. / Madl, T. / Erdmann, R. / Schliebs, W. / Wilmanns, M. / Sattler, M. |
|---|
 引用 引用 |  ジャーナル: Embo J. / 年: 2009 ジャーナル: Embo J. / 年: 2009
タイトル: Structural Basis for Competitive Interactions of Pex14 with the Import Receptors Pex5 and Pex19.
著者: Neufeld, C. / Filipp, F.V. / Simon, B. / Neuhaus, A. / Schueller, N. / David, C. / Kooshapur, H. / Madl, T. / Erdmann, R. / Schliebs, W. / Wilmanns, M. / Sattler, M. |
|---|
| 履歴 | | 登録 | 2009年1月9日 | 登録サイト: PDBE / 処理サイト: PDBE |
|---|
| 改定 1.0 | 2009年2月17日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2013年5月8日 | Group: Derived calculations / Other ...Derived calculations / Other / Refinement description / Structure summary / Version format compliance |
|---|
| 改定 1.2 | 2024年5月15日 | Group: Data collection / Database references / Other
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_database_status / pdbx_nmr_software
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_database_status.status_code_mr / _pdbx_nmr_software.name |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 HOMO SAPIENS (ヒト)
HOMO SAPIENS (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Embo J. / 年: 2009
ジャーナル: Embo J. / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2w85.cif.gz
2w85.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2w85.ent.gz
pdb2w85.ent.gz PDB形式
PDB形式 2w85.json.gz
2w85.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2w85_validation.pdf.gz
2w85_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2w85_full_validation.pdf.gz
2w85_full_validation.pdf.gz 2w85_validation.xml.gz
2w85_validation.xml.gz 2w85_validation.cif.gz
2w85_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/w8/2w85
https://data.pdbj.org/pub/pdb/validation_reports/w8/2w85 ftp://data.pdbj.org/pub/pdb/validation_reports/w8/2w85
ftp://data.pdbj.org/pub/pdb/validation_reports/w8/2w85 リンク
リンク 集合体
集合体
 要素
要素 HOMO SAPIENS (ヒト) / プラスミド: PETM11_MOD / 発現宿主:
HOMO SAPIENS (ヒト) / プラスミド: PETM11_MOD / 発現宿主: 
 HOMO SAPIENS (ヒト) / 参照: UniProt: P40855
HOMO SAPIENS (ヒト) / 参照: UniProt: P40855 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj
 HNCA
HNCA