登録情報 データベース : PDB / ID : 2mivタイトル NMR studies of N2-guanine adducts derived from the tumorigen dibenzo[a,l]pyrene in DNA: Impact of adduct stereochemistry, size, and local DNA structure on solution conformations DNA_(5'-D(*CP*CP*AP*TP*CP*GP*CP*TP*AP*CP*C)-3')DNA_(5'-D(*GP*GP*TP*AP*GP*GP*AP*TP*GP*G)-3') キーワード / / / 機能・相同性 / / 手法 / Model details closest to the average, model1 データ登録者 Rodriguez, F.A. / Liu, Z. / Lin, C.H. / Ding, S. / Cai, Y. / Kolbanovskiy, A. / Kolbanovskiy, M. / Amin, S. / Broyde, S. / Geacintov, N.E. ジャーナル : Biochemistry / 年 : 2014タイトル : Nuclear Magnetic Resonance Studies of an N(2)-Guanine Adduct Derived from the Tumorigen Dibenzo[a,l]pyrene in DNA: Impact of Adduct Stereochemistry, Size, and Local DNA Sequence on Solution Conformations.著者 : Rodriguez, F.A. / Liu, Z. / Lin, C.H. / Ding, S. / Cai, Y. / Kolbanovskiy, A. / Kolbanovskiy, M. / Amin, S. / Broyde, S. / Geacintov, N.E. 履歴 登録 2013年12月20日 登録サイト / 処理サイト 改定 1.0 2014年4月2日 Provider / タイプ 改定 1.1 2014年4月9日 Group 改定 1.2 2024年5月1日 Group / Database references / Derived calculationsカテゴリ chem_comp_atom / chem_comp_bond ... chem_comp_atom / chem_comp_bond / database_2 / pdbx_nmr_software / pdbx_nmr_spectrometer / struct_conn / struct_site Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 2014
ジャーナル: Biochemistry / 年: 2014 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2miv.cif.gz
2miv.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2miv.ent.gz
pdb2miv.ent.gz PDB形式
PDB形式 2miv.json.gz
2miv.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2miv_validation.pdf.gz
2miv_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2miv_full_validation.pdf.gz
2miv_full_validation.pdf.gz 2miv_validation.xml.gz
2miv_validation.xml.gz 2miv_validation.cif.gz
2miv_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/mi/2miv
https://data.pdbj.org/pub/pdb/validation_reports/mi/2miv ftp://data.pdbj.org/pub/pdb/validation_reports/mi/2miv
ftp://data.pdbj.org/pub/pdb/validation_reports/mi/2miv リンク
リンク 集合体
集合体
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー



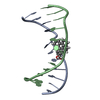





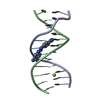

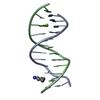


 PDBj
PDBj







































 Amber
Amber