| 登録情報 | データベース: PDB / ID: 2m7h
|
|---|
| タイトル | The C-terminal Region of Disintegrin Modulate its 3D Conformation and Cooperate with RGD Loop in Regulating Integrin alpha-IIb beta-3 Recognition |
|---|
 要素 要素 | Zinc metalloproteinase/disintegrin |
|---|
 キーワード キーワード | HYDROLASE / Rhodostomin muant / RGD motif / C-terminal region mutations / integrin alpha-IIb beta-3 |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
加水分解酵素; プロテアーゼ; ペプチド結合加水分解酵素; その他のペプチターゼ / metalloendopeptidase activity / toxin activity / proteolysis / extracellular region / metal ion binding / plasma membrane類似検索 - 分子機能 Disintegrin domain / Echistatin / Disintegrin, conserved site / Disintegrins signature. / Peptidase M12B, propeptide / Reprolysin family propeptide / Reprolysin domain, adamalysin-type / Reprolysin (M12B) family zinc metalloprotease / Disintegrin / Disintegrin domain profile. ...Disintegrin domain / Echistatin / Disintegrin, conserved site / Disintegrins signature. / Peptidase M12B, propeptide / Reprolysin family propeptide / Reprolysin domain, adamalysin-type / Reprolysin (M12B) family zinc metalloprotease / Disintegrin / Disintegrin domain profile. / Homologues of snake disintegrins / Disintegrin domain / Disintegrin domain superfamily / Peptidase M12B, ADAM/reprolysin / ADAM type metalloprotease domain profile. / Metallopeptidase, catalytic domain superfamily / Neutral zinc metallopeptidases, zinc-binding region signature. / Few Secondary Structures / Irregular類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Calloselasma rhodostoma (ヘビ) Calloselasma rhodostoma (ヘビ) |
|---|
| 手法 | 溶液NMR / DGSA-distance geometry simulated annealing |
|---|
| Model details | closest to the average, model8 |
|---|
 データ登録者 データ登録者 | Chuang, W. / Chang, Y. |
|---|
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Published
タイトル: The C-terminal Region of Disintegrin Modulate its 3D Conformation and Cooperate with RGD Loop in Regulating Recognitions of Integrins
著者: Chuang, W. / Chang, Y. / Shiu, J. / Chen, C. / Chen, Y. |
|---|
| 履歴 | | 登録 | 2013年4月22日 | 登録サイト: BMRB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2013年5月22日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年11月13日 | Group: Data collection / Database references / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_nmr_software / pdbx_nmr_spectrometer / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _struct_ref_seq_dif.details |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Calloselasma rhodostoma (ヘビ)
Calloselasma rhodostoma (ヘビ) データ登録者
データ登録者 引用
引用 ジャーナル: To be Published
ジャーナル: To be Published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2m7h.cif.gz
2m7h.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2m7h.ent.gz
pdb2m7h.ent.gz PDB形式
PDB形式 2m7h.json.gz
2m7h.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2m7h_validation.pdf.gz
2m7h_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2m7h_full_validation.pdf.gz
2m7h_full_validation.pdf.gz 2m7h_validation.xml.gz
2m7h_validation.xml.gz 2m7h_validation.cif.gz
2m7h_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/m7/2m7h
https://data.pdbj.org/pub/pdb/validation_reports/m7/2m7h ftp://data.pdbj.org/pub/pdb/validation_reports/m7/2m7h
ftp://data.pdbj.org/pub/pdb/validation_reports/m7/2m7h リンク
リンク 集合体
集合体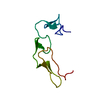
 要素
要素 Calloselasma rhodostoma (ヘビ) / 遺伝子: RHOD / 発現宿主:
Calloselasma rhodostoma (ヘビ) / 遺伝子: RHOD / 発現宿主:  Komagataella pastoris (菌類)
Komagataella pastoris (菌類) 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj


 HSQC
HSQC