+ データを開く
データを開く
- 基本情報
基本情報
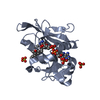
| 登録情報 | データベース: PDB / ID: 2lxn | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Solution NMR structure of glutamine amido transferase subunit of gaunosine monophosphate synthetase from Methanocaldococcus jannaschii | ||||||
 要素 要素 | GMP synthase [glutamine-hydrolyzing] subunit A | ||||||
 キーワード キーワード | LIGASE / Glutamine amidotransferase / Ammonia channeling / De-novo purine nucleotide biosynthesis / Solution NMR structure / Methanocaldococcus jannaschii | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報GMP synthase (glutamine-hydrolyzing) activity / GMP synthase (glutamine-hydrolysing) / ATP binding 類似検索 - 分子機能 | ||||||
| 生物種 |   Methanocaldococcus jannaschii (メタン生成菌) Methanocaldococcus jannaschii (メタン生成菌) | ||||||
| 手法 | 溶液NMR / na | ||||||
| Model details | Target Function, model1 | ||||||
 データ登録者 データ登録者 | Ali, R. / Kumar, S. / Balaram, H. / Sarma, S.P. | ||||||
 引用 引用 |  ジャーナル: Biomol.Nmr Assign. / 年: 2012 ジャーナル: Biomol.Nmr Assign. / 年: 2012タイトル: 1H, 13C, 15N assignment and secondary structure determination of glutamine amido transferase subunit of gaunosine monophosphate synthetase from Methanocaldococcus jannaschii 著者: Ali, R. / Kumar, S. / Balaram, H. / Sarma, S.P. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2lxn.cif.gz 2lxn.cif.gz | 1.3 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2lxn.ent.gz pdb2lxn.ent.gz | 1.1 MB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2lxn.json.gz 2lxn.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2lxn_validation.pdf.gz 2lxn_validation.pdf.gz | 469.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2lxn_full_validation.pdf.gz 2lxn_full_validation.pdf.gz | 696.9 KB | 表示 | |
| XML形式データ |  2lxn_validation.xml.gz 2lxn_validation.xml.gz | 100.3 KB | 表示 | |
| CIF形式データ |  2lxn_validation.cif.gz 2lxn_validation.cif.gz | 107.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/lx/2lxn https://data.pdbj.org/pub/pdb/validation_reports/lx/2lxn ftp://data.pdbj.org/pub/pdb/validation_reports/lx/2lxn ftp://data.pdbj.org/pub/pdb/validation_reports/lx/2lxn | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
|
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質 | 分子量: 21051.250 Da / 分子数: 1 / 由来タイプ: 組換発現 由来: (組換発現)   Methanocaldococcus jannaschii (メタン生成菌) Methanocaldococcus jannaschii (メタン生成菌)株: ATCC 43067 / DSM 2661 / JAL-1 / JCM 10045 / NBRC 100440 遺伝子: guaAA, MJ1575 / 発現宿主:  参照: UniProt: Q58970, GMP synthase (glutamine-hydrolysing) |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 試料状態 | pH: 7 / 圧: ambient / 温度: 303 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Bruker Avance / 製造業者: Bruker / モデル: AVANCE / 磁場強度: 700 MHz |
|---|
- 解析
解析
| NMR software |
|
|---|
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj

 HSQC
HSQC