| 登録情報 | データベース: PDB / ID: 2lk1
|
|---|
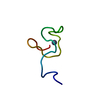
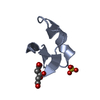
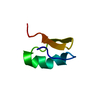
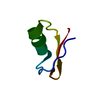
| タイトル | Solution structure and binding studies of the RanBP2-type zinc finger of RBM5 |
|---|
 要素 要素 | RNA-binding protein 5 |
|---|
 キーワード キーワード | RNA BINDING PROTEIN / Zinc finger |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
regulation of alternative mRNA splicing, via spliceosome / spliceosomal complex assembly / RNA processing / mRNA Splicing - Major Pathway / spliceosomal complex / mRNA splicing, via spliceosome / positive regulation of apoptotic process / negative regulation of cell population proliferation / mRNA binding / intracellular membrane-bounded organelle ...regulation of alternative mRNA splicing, via spliceosome / spliceosomal complex assembly / RNA processing / mRNA Splicing - Major Pathway / spliceosomal complex / mRNA splicing, via spliceosome / positive regulation of apoptotic process / negative regulation of cell population proliferation / mRNA binding / intracellular membrane-bounded organelle / apoptotic process / DNA binding / RNA binding / zinc ion binding / nucleoplasm / nucleus / cytosol類似検索 - 分子機能 RNA-binding protein 5, RNA recognition motif 1 / RNA-binding protein 5, RNA recognition motif 2 / OCRE domain / OCRE domain / G-patch domain / G-patch domain profile. / G-patch domain / glycine rich nucleic binding domain / Zinc finger domain / Zn-finger in Ran binding protein and others ...RNA-binding protein 5, RNA recognition motif 1 / RNA-binding protein 5, RNA recognition motif 2 / OCRE domain / OCRE domain / G-patch domain / G-patch domain profile. / G-patch domain / glycine rich nucleic binding domain / Zinc finger domain / Zn-finger in Ran binding protein and others / Zinc finger RanBP2 type profile. / Zinc finger, RanBP2-type superfamily / Zinc finger RanBP2-type signature. / Zinc finger, RanBP2-type / Zinc finger C2H2 type domain profile. / Zinc finger C2H2-type / RNA recognition motif / RNA recognition motif / Eukaryotic RNA Recognition Motif (RRM) profile. / RNA recognition motif domain / RNA-binding domain superfamily / Nucleotide-binding alpha-beta plait domain superfamily類似検索 - ドメイン・相同性 9,10-dioxo-9,10-dihydroanthracene-2-sulfonic acid / RNA-binding protein 5類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing, Energy minimization |
|---|
| Model details | closest to the average, model 5 |
|---|
 データ登録者 データ登録者 | Farina, B. / Pellecchia, M. |
|---|
 引用 引用 |  ジャーナル: Chembiochem / 年: 2011 ジャーナル: Chembiochem / 年: 2011
タイトル: Targeting Zinc Finger Domains with Small Molecules: Solution Structure and Binding Studies of the RanBP2-Type Zinc Finger of RBM5.
著者: Farina, B. / Fattorusso, R. / Pellecchia, M. |
|---|
| 履歴 | | 登録 | 2011年9月30日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2011年12月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2024年11月27日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_nmr_software / pdbx_nmr_spectrometer / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _pdbx_nmr_spectrometer.model / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.pdbx_leaving_atom_flag / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_seq_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Chembiochem / 年: 2011
ジャーナル: Chembiochem / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2lk1.cif.gz
2lk1.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2lk1.ent.gz
pdb2lk1.ent.gz PDB形式
PDB形式 2lk1.json.gz
2lk1.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2lk1_validation.pdf.gz
2lk1_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2lk1_full_validation.pdf.gz
2lk1_full_validation.pdf.gz 2lk1_validation.xml.gz
2lk1_validation.xml.gz 2lk1_validation.cif.gz
2lk1_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/lk/2lk1
https://data.pdbj.org/pub/pdb/validation_reports/lk/2lk1 ftp://data.pdbj.org/pub/pdb/validation_reports/lk/2lk1
ftp://data.pdbj.org/pub/pdb/validation_reports/lk/2lk1 リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 参照: UniProt: P52756
Homo sapiens (ヒト) / 参照: UniProt: P52756 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー





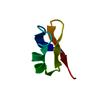

 PDBj
PDBj











 CYANA
CYANA