+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2l8f | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Structure of a 4X4 Nucleotide RNA Internal Loop from an R2 Retrotransposon | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | RNA / RNA motif / internal loop / 3RRs | ||||||
| 機能・相同性 | RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
| Model details | lowest constraint energy, model 1 | ||||||
 データ登録者 データ登録者 | Lerman, Y. / Kennedy, S.D. / Turner, D.H. | ||||||
 引用 引用 |  ジャーナル: Rna / 年: 2011 ジャーナル: Rna / 年: 2011タイトル: NMR structure of a 4 x 4 nucleotide RNA internal loop from an R2 retrotransposon: Identification of a three purine-purine sheared pair motif and comparison to MC-SYM predictions. 著者: Lerman, Y.V. / Kennedy, S.D. / Shankar, N. / Parisien, M. / Major, F. / Turner, D.H. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2l8f.cif.gz 2l8f.cif.gz | 142.5 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2l8f.ent.gz pdb2l8f.ent.gz | 117.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2l8f.json.gz 2l8f.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2l8f_validation.pdf.gz 2l8f_validation.pdf.gz | 426.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2l8f_full_validation.pdf.gz 2l8f_full_validation.pdf.gz | 526.1 KB | 表示 | |
| XML形式データ |  2l8f_validation.xml.gz 2l8f_validation.xml.gz | 7.8 KB | 表示 | |
| CIF形式データ |  2l8f_validation.cif.gz 2l8f_validation.cif.gz | 11.7 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/l8/2l8f https://data.pdbj.org/pub/pdb/validation_reports/l8/2l8f ftp://data.pdbj.org/pub/pdb/validation_reports/l8/2l8f ftp://data.pdbj.org/pub/pdb/validation_reports/l8/2l8f | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 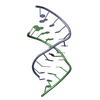
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 3522.154 Da / 分子数: 1 / 由来タイプ: 合成 |
|---|---|
| #2: RNA鎖 | 分子量: 3545.194 Da / 分子数: 1 / 由来タイプ: 合成 |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| |||||||||||||||||||||||||||||||||
| 試料状態 |
|
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 | ||||||||||||||||||||||||||||||||
| NMR constraints | NOE constraints total: 215 / NOE intraresidue total count: 79 / NOE long range total count: 40 / NOE sequential total count: 136 | ||||||||||||||||||||||||||||||||
| 代表構造 | 選択基準: lowest constraint energy | ||||||||||||||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: no restraint violations; low constraint energy 計算したコンフォーマーの数: 40 / 登録したコンフォーマーの数: 10 / Maximum lower distance constraint violation: 0 Å / Maximum upper distance constraint violation: 0.18 Å | ||||||||||||||||||||||||||||||||
| NMR ensemble rms | Distance rms dev: 0.003 Å |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj





























 HSQC
HSQC