+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2l8e | ||||||
|---|---|---|---|---|---|---|---|
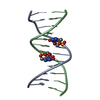
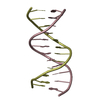
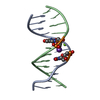
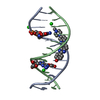
| タイトル | Solution NMR structure of FCS domain of Human Polyhomeotic Homolog 1 (HPH1) | ||||||
 要素 要素 | Polyhomeotic-like protein 1 | ||||||
 キーワード キーワード | DNA BINDING PROTEIN | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報: / PRC1 complex / multicellular organism development / PcG protein complex / negative regulation of G0 to G1 transition / SUMOylation of DNA methylation proteins / SUMOylation of RNA binding proteins / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / Transcriptional Regulation by E2F6 / SUMOylation of DNA damage response and repair proteins ...: / PRC1 complex / multicellular organism development / PcG protein complex / negative regulation of G0 to G1 transition / SUMOylation of DNA methylation proteins / SUMOylation of RNA binding proteins / RUNX1 interacts with co-factors whose precise effect on RUNX1 targets is not known / Transcriptional Regulation by E2F6 / SUMOylation of DNA damage response and repair proteins / SUMOylation of chromatin organization proteins / SUMOylation of transcription cofactors / Regulation of PTEN gene transcription / histone binding / Oxidative Stress Induced Senescence / negative regulation of DNA-templated transcription / chromatin binding / DNA binding / zinc ion binding / nucleoplasm / nucleus 類似検索 - 分子機能 | ||||||
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 | 溶液NMR / DGSA-distance geometry simulated annealing | ||||||
| Model details | lowest energy, model 1 | ||||||
 データ登録者 データ登録者 | Ilangovan, U. / Kim, C. | ||||||
 引用 引用 |  ジャーナル: To be Published ジャーナル: To be Publishedタイトル: 1H, 15N and 13C assigned chemical shifts of FCS domain from human polyhomeotic homolog 1 著者: Wang, R. / Ilangovan, U. / Kim, C. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2l8e.cif.gz 2l8e.cif.gz | 333.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2l8e.ent.gz pdb2l8e.ent.gz | 279.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2l8e.json.gz 2l8e.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2l8e_validation.pdf.gz 2l8e_validation.pdf.gz | 398.6 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2l8e_full_validation.pdf.gz 2l8e_full_validation.pdf.gz | 511.5 KB | 表示 | |
| XML形式データ |  2l8e_validation.xml.gz 2l8e_validation.xml.gz | 17.8 KB | 表示 | |
| CIF形式データ |  2l8e_validation.cif.gz 2l8e_validation.cif.gz | 29 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/l8/2l8e https://data.pdbj.org/pub/pdb/validation_reports/l8/2l8e ftp://data.pdbj.org/pub/pdb/validation_reports/l8/2l8e ftp://data.pdbj.org/pub/pdb/validation_reports/l8/2l8e | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 5490.305 Da / 分子数: 1 / 断片: FCS-type zinc finger domain residues 783-828 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 遺伝子: PHC1, EDR1, PH1 / 発現宿主: Homo sapiens (ヒト) / 遺伝子: PHC1, EDR1, PH1 / 発現宿主:  |
|---|---|
| #2: 化合物 | ChemComp-ZN / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
|
- 試料調製
試料調製
| 詳細 |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料 |
| ||||||||||||||||
| 試料状態 | イオン強度: 0.050 / pH: 6.0 / 圧: ambient / 温度: 300 K |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: DGSA-distance geometry simulated annealing / ソフトェア番号: 1 | |||||||||||||||
| 代表構造 | 選択基準: lowest energy | |||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 200 / 登録したコンフォーマーの数: 20 |
 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj










 HSQC
HSQC