| 登録情報 | データベース: PDB / ID: 2k84
|
|---|
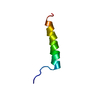
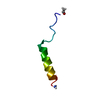
| タイトル | Solution Structure of the equine infectious anemia virus p9 GAG protein |
|---|
 要素 要素 | P9 |
|---|
 キーワード キーワード | VIRAL PROTEIN / polypeptide / Capsid protein / Core protein / Host-virus interaction / Metal-binding / Viral matrix protein / Viral nucleoprotein / Virion / Zinc / Zinc-finger |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
viral budding via host ESCRT complex / viral nucleocapsid / structural constituent of virion / nucleic acid binding / zinc ion binding類似検索 - 分子機能 Gag protein p15 / Gag protein p15 / : / Matrix protein, lentiviral and alpha-retroviral, N-terminal / Retroviral nucleocapsid Gag protein p24, C-terminal domain / Gag protein p24 C-terminal domain / Retrovirus capsid, C-terminal / Retroviral matrix protein / Retrovirus capsid, N-terminal / zinc finger ...Gag protein p15 / Gag protein p15 / : / Matrix protein, lentiviral and alpha-retroviral, N-terminal / Retroviral nucleocapsid Gag protein p24, C-terminal domain / Gag protein p24 C-terminal domain / Retrovirus capsid, C-terminal / Retroviral matrix protein / Retrovirus capsid, N-terminal / zinc finger / Zinc knuckle / Zinc finger, CCHC-type superfamily / Zinc finger, CCHC-type / Zinc finger CCHC-type profile.類似検索 - ドメイン・相同性 |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
| Model type details | minimized average |
|---|
 データ登録者 データ登録者 | Sharma, A. / Bruns, K. / Roeder, R. / Henklein, P. / Votteler, J. / Wray, V. / Sharma, U. |
|---|
 引用 引用 |  ジャーナル: Bmc Struct.Biol. / 年: 2009 ジャーナル: Bmc Struct.Biol. / 年: 2009
タイトル: Solution structure of the Equine Infectious Anemia Virus p9 protein: a rationalization of its different ALIX binding requirements compared to the analogous HIV-p6 protein
著者: Sharma, A. / Bruns, K. / Roder, R. / Henklein, P. / Votteler, J. / Wray, V. / Schubert, U. |
|---|
| 履歴 | | 登録 | 2008年9月2日 | 登録サイト: BMRB / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2009年9月8日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2022年3月16日 | Group: Database references / Derived calculations
カテゴリ: database_2 / pdbx_struct_assembly / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
| 改定 1.3 | 2024年5月29日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 引用
引用 ジャーナル: Bmc Struct.Biol. / 年: 2009
ジャーナル: Bmc Struct.Biol. / 年: 2009 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2k84.cif.gz
2k84.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2k84.ent.gz
pdb2k84.ent.gz PDB形式
PDB形式 2k84.json.gz
2k84.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2k84_validation.pdf.gz
2k84_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2k84_full_validation.pdf.gz
2k84_full_validation.pdf.gz 2k84_validation.xml.gz
2k84_validation.xml.gz 2k84_validation.cif.gz
2k84_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/k8/2k84
https://data.pdbj.org/pub/pdb/validation_reports/k8/2k84 ftp://data.pdbj.org/pub/pdb/validation_reports/k8/2k84
ftp://data.pdbj.org/pub/pdb/validation_reports/k8/2k84 リンク
リンク 集合体
集合体
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj
