| 登録情報 | データベース: PDB / ID: 2k0u
|
|---|
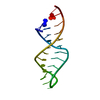
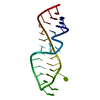
| タイトル | High Resolution Solution NMR Structures of Oxaliplatin-DNA Adduct |
|---|
 要素 要素 | - DNA (5'-D(*DCP*DCP*DTP*DCP*DTP*DGP*DGP*DTP*DCP*DTP*DCP*DC)-3')
- DNA (5'-D(*DGP*DGP*DAP*DGP*DAP*DCP*DCP*DAP*DGP*DAP*DGP*DG)-3')
|
|---|
 キーワード キーワード | DNA / Oxaliplatin-DNA Adduct |
|---|
| 機能・相同性 | CYCLOHEXANE-1(R),2(R)-DIAMINE-PLATINUM(II) / DNA / DNA (> 10) 機能・相同性情報 機能・相同性情報 |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
| Model type details | minimized average |
|---|
 データ登録者 データ登録者 | Bhattacharyya, D. / King, C.L. / Chaney, S.G. / Campbell, S.L. |
|---|
 引用 引用 |  ジャーナル: Plos One / 年: 2011 ジャーナル: Plos One / 年: 2011
タイトル: Flanking Bases Influence the Nature of DNA Distortion by Platinum 1,2-Intrastrand (GG) Cross-Links.
著者: Bhattacharyya, D. / Ramachandran, S. / Sharma, S. / Pathmasiri, W. / King, C.L. / Baskerville-Abraham, I. / Boysen, G. / Swenberg, J.A. / Campbell, S.L. / Dokholyan, N.V. / Chaney, S.G. |
|---|
| 履歴 | | 登録 | 2008年2月15日 | 登録サイト: BMRB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2009年2月3日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年8月31日 | Group: Database references |
|---|
| 改定 1.3 | 2024年5月1日 | Group: Data collection / Database references / Derived calculations
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / struct_ref_seq / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq.db_align_beg / _struct_ref_seq.db_align_end / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 引用
引用 ジャーナル: Plos One / 年: 2011
ジャーナル: Plos One / 年: 2011 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2k0u.cif.gz
2k0u.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2k0u.ent.gz
pdb2k0u.ent.gz PDB形式
PDB形式 2k0u.json.gz
2k0u.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2k0u_validation.pdf.gz
2k0u_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2k0u_full_validation.pdf.gz
2k0u_full_validation.pdf.gz 2k0u_validation.xml.gz
2k0u_validation.xml.gz 2k0u_validation.cif.gz
2k0u_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/k0/2k0u
https://data.pdbj.org/pub/pdb/validation_reports/k0/2k0u ftp://data.pdbj.org/pub/pdb/validation_reports/k0/2k0u
ftp://data.pdbj.org/pub/pdb/validation_reports/k0/2k0u リンク
リンク 集合体
集合体
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj







































 HSQC
HSQC