登録情報 データベース : PDB / ID : 2ejsタイトル Solution structure of RUH-076, a human CUE domain Autocrine motility factor receptor, isoform 2 キーワード / / / / / / / / 機能・相同性 分子機能 ドメイン・相同性 構成要素
/ / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / / 生物種 Homo sapiens (ヒト)手法 / データ登録者 Ruhul Momen, A.Z.M. / Kitasaka, S. / Hirota, H. / Hayashi, F. / Yokoyama, S. / RIKEN Structural Genomics/Proteomics Initiative (RSGI) ジャーナル : to be published タイトル : Solution structure of RUH-076, a human CUE domain著者 : Ruhul Momen, A.Z.M. / Kitasaka, S. / Hirota, H. / Hayashi, F. / Yokoyama, S. 履歴 登録 2007年3月20日 登録サイト / 処理サイト 改定 1.0 2007年9月25日 Provider / タイプ 改定 1.1 2011年7月13日 Group 改定 1.2 2022年3月9日 Group / Database references / Derived calculationsカテゴリ database_2 / pdbx_nmr_software ... database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref_seq_dif Item _database_2.pdbx_DOI / _database_2.pdbx_database_accession ... _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _struct_ref_seq_dif.details 改定 1.3 2024年5月29日 Group / カテゴリ / chem_comp_bond
すべて表示 表示を減らす
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: to be published
ジャーナル: to be published 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2ejs.cif.gz
2ejs.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2ejs.ent.gz
pdb2ejs.ent.gz PDB形式
PDB形式 2ejs.json.gz
2ejs.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2ejs_validation.pdf.gz
2ejs_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2ejs_full_validation.pdf.gz
2ejs_full_validation.pdf.gz 2ejs_validation.xml.gz
2ejs_validation.xml.gz 2ejs_validation.cif.gz
2ejs_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/ej/2ejs
https://data.pdbj.org/pub/pdb/validation_reports/ej/2ejs ftp://data.pdbj.org/pub/pdb/validation_reports/ej/2ejs
ftp://data.pdbj.org/pub/pdb/validation_reports/ej/2ejs リンク
リンク 集合体
集合体
 要素
要素 Homo sapiens (ヒト) / 解説: Cell-free protein synthesis / プラスミド: P060904-10
Homo sapiens (ヒト) / 解説: Cell-free protein synthesis / プラスミド: P060904-10 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー








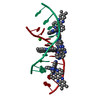
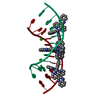

 PDBj
PDBj




 NMRPipe
NMRPipe