| 登録情報 | データベース: PDB / ID: 2e4h
|
|---|
| タイトル | Solution structure of cytoskeletal protein in complex with tubulin tail |
|---|
 要素 要素 | - Restin
- Tubulin alpha-ubiquitous chain
|
|---|
 キーワード キーワード | STRUCTURAL PROTEIN / cytoskeleton / CAP-Gly / complex structure / tubulin / solution structure / CLIP |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Post-chaperonin tubulin folding pathway / Cilium Assembly / cytoskeleton-dependent intracellular transport / Microtubule-dependent trafficking of connexons from Golgi to the plasma membrane / Carboxyterminal post-translational modifications of tubulin / Intraflagellar transport / Sealing of the nuclear envelope (NE) by ESCRT-III / Formation of tubulin folding intermediates by CCT/TriC / Gap junction assembly / microtubule bundle formation ...Post-chaperonin tubulin folding pathway / Cilium Assembly / cytoskeleton-dependent intracellular transport / Microtubule-dependent trafficking of connexons from Golgi to the plasma membrane / Carboxyterminal post-translational modifications of tubulin / Intraflagellar transport / Sealing of the nuclear envelope (NE) by ESCRT-III / Formation of tubulin folding intermediates by CCT/TriC / Gap junction assembly / microtubule bundle formation / microtubule plus-end binding / Kinesins / Assembly and cell surface presentation of NMDA receptors / COPI-independent Golgi-to-ER retrograde traffic / Signaling by LTK in cancer / intermediate filament / COPI-dependent Golgi-to-ER retrograde traffic / Recycling pathway of L1 / RHOH GTPase cycle / RHO GTPases activate IQGAPs / microtubule-based process / Hedgehog 'off' state / Activation of AMPK downstream of NMDARs / COPI-mediated anterograde transport / cytoplasmic microtubule / ruffle / Amplification of signal from unattached kinetochores via a MAD2 inhibitory signal / cytoplasmic microtubule organization / positive regulation of microtubule polymerization / Mitotic Prometaphase / EML4 and NUDC in mitotic spindle formation / MHC class II antigen presentation / cellular response to interleukin-4 / tubulin binding / Recruitment of NuMA to mitotic centrosomes / HSP90 chaperone cycle for steroid hormone receptors (SHR) in the presence of ligand / cytoplasmic vesicle membrane / Resolution of Sister Chromatid Cohesion / Translocation of SLC2A4 (GLUT4) to the plasma membrane / RHO GTPases Activate Formins / PKR-mediated signaling / kinetochore / structural constituent of cytoskeleton / Aggrephagy / microtubule cytoskeleton organization / The role of GTSE1 in G2/M progression after G2 checkpoint / Separation of Sister Chromatids / HCMV Early Events / mitotic cell cycle / double-stranded RNA binding / microtubule cytoskeleton / cell cortex / microtubule binding / 加水分解酵素; 酸無水物に作用; GTPに作用・細胞または細胞小器官の運動に関与 / microtubule / cilium / cell division / GTPase activity / ubiquitin protein ligase binding / centrosome / GTP binding / structural molecule activity / zinc ion binding / identical protein binding / nucleus / plasma membrane / cytosol / cytoplasm類似検索 - 分子機能 CLIP1, zinc knuckle / CLIP1 zinc knuckle / CAP Gly-rich-like domain / CAP-Gly domain signature. / CAP Gly-rich domain / CAP Gly-rich domain superfamily / CAP-Gly domain / CAP-Gly domain profile. / CAP_GLY / Alpha tubulin ...CLIP1, zinc knuckle / CLIP1 zinc knuckle / CAP Gly-rich-like domain / CAP-Gly domain signature. / CAP Gly-rich domain / CAP Gly-rich domain superfamily / CAP-Gly domain / CAP-Gly domain profile. / CAP_GLY / Alpha tubulin / Tubulin / Tubulin, C-terminal / Tubulin C-terminal domain / Tubulin, conserved site / Tubulin subunits alpha, beta, and gamma signature. / Tubulin/FtsZ family, C-terminal domain / Tubulin/FtsZ-like, C-terminal domain / Tubulin/FtsZ, C-terminal / Tubulin/FtsZ, 2-layer sandwich domain / Tubulin/FtsZ family, GTPase domain / Tubulin/FtsZ family, GTPase domain / Tubulin/FtsZ, GTPase domain / Tubulin/FtsZ, GTPase domain superfamily / SH3 type barrels. / Roll / Mainly Beta類似検索 - ドメイン・相同性 CAP-Gly domain-containing linker protein 1 / Tubulin alpha-1B chain類似検索 - 構成要素 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Mishima, M. / Hakoshima, T. |
|---|
 引用 引用 |  ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007
タイトル: Structural basis for tubulin recognition by cytoplasmic linker protein 170 and its autoinhibition
著者: Mishima, M. / Maesaki, R. / Kasa, M. / Watanabe, T. / Fukata, M. / Kaibuchi, K. / Hakoshima, T. |
|---|
| 履歴 | | 登録 | 2006年12月8日 | 登録サイト: PDBJ / 処理サイト: PDBJ |
|---|
| 改定 1.0 | 2007年8月28日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2022年3月9日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_spectrometer ...database_2 / pdbx_nmr_spectrometer / pdbx_struct_assembly / pdbx_struct_oper_list
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_spectrometer.model |
|---|
| 改定 1.3 | 2024年5月29日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) データ登録者
データ登録者 引用
引用 ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007
ジャーナル: Proc.Natl.Acad.Sci.Usa / 年: 2007 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 2e4h.cif.gz
2e4h.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb2e4h.ent.gz
pdb2e4h.ent.gz PDB形式
PDB形式 2e4h.json.gz
2e4h.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 2e4h_validation.pdf.gz
2e4h_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 2e4h_full_validation.pdf.gz
2e4h_full_validation.pdf.gz 2e4h_validation.xml.gz
2e4h_validation.xml.gz 2e4h_validation.cif.gz
2e4h_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/e4/2e4h
https://data.pdbj.org/pub/pdb/validation_reports/e4/2e4h ftp://data.pdbj.org/pub/pdb/validation_reports/e4/2e4h
ftp://data.pdbj.org/pub/pdb/validation_reports/e4/2e4h リンク
リンク 集合体
集合体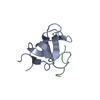
 要素
要素 Homo sapiens (ヒト) / 遺伝子: CLIP-170 / プラスミド: pGEX4T / 生物種 (発現宿主): Escherichia coli / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: CLIP-170 / プラスミド: pGEX4T / 生物種 (発現宿主): Escherichia coli / 発現宿主: 
 Homo sapiens (ヒト) / 遺伝子: a3-tubulin / プラスミド: pGEX6P / 発現宿主:
Homo sapiens (ヒト) / 遺伝子: a3-tubulin / プラスミド: pGEX6P / 発現宿主: 
 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












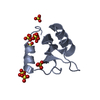


 PDBj
PDBj



























 NMRPipe
NMRPipe