+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 2bao | ||||||
|---|---|---|---|---|---|---|---|
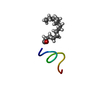
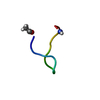
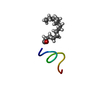
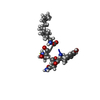
| タイトル | Solution NMR structure of the myristoylated N-terminal fragment of Arf6 | ||||||
 要素 要素 | ADP-ribosylation factor 6 | ||||||
 キーワード キーワード | SIGNALING PROTEIN / Myristoyl / N-terminal / Arf6 | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報erythrocyte apoptotic process / maintenance of postsynaptic density structure / protein localization to cleavage furrow / positive regulation of mitotic cytokinetic process / negative regulation of dendrite development / establishment of epithelial cell polarity / regulation of dendritic spine development / regulation of Rac protein signal transduction / protein localization to endosome / negative regulation of protein localization to cell surface ...erythrocyte apoptotic process / maintenance of postsynaptic density structure / protein localization to cleavage furrow / positive regulation of mitotic cytokinetic process / negative regulation of dendrite development / establishment of epithelial cell polarity / regulation of dendritic spine development / regulation of Rac protein signal transduction / protein localization to endosome / negative regulation of protein localization to cell surface / negative regulation of receptor-mediated endocytosis / ruffle assembly / positive regulation of keratinocyte migration / positive regulation of focal adhesion disassembly / regulation of filopodium assembly / MET receptor recycling / endocytic recycling / thioesterase binding / Flemming body / filopodium membrane / TBC/RABGAPs / protein localization to cell surface / cortical actin cytoskeleton organization / hepatocyte apoptotic process / positive regulation of actin filament polymerization / cleavage furrow / synaptic vesicle endocytosis / endocytic vesicle / regulation of presynapse assembly / vesicle-mediated transport / ruffle / signaling adaptor activity / small monomeric GTPase / protein localization to plasma membrane / positive regulation of protein secretion / positive regulation of protein localization to plasma membrane / intracellular protein transport / liver development / positive regulation of neuron projection development / cellular response to nerve growth factor stimulus / recycling endosome membrane / GDP binding / Clathrin-mediated endocytosis / nervous system development / presynapse / G protein activity / midbody / cell cortex / early endosome membrane / cell differentiation / postsynapse / cell adhesion / endosome / cell division / focal adhesion / GTPase activity / GTP binding / glutamatergic synapse / Golgi apparatus / extracellular exosome / membrane / plasma membrane / cytosol / cytoplasm 類似検索 - 分子機能 | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | Gizachew, D. | ||||||
 引用 引用 |  ジャーナル: Febs Lett. / 年: 2006 ジャーナル: Febs Lett. / 年: 2006タイトル: NMR structural studies of the myristoylated N-terminus of ADP ribosylation factor 6 (Arf6). 著者: Gizachew, D. / Oswald, R. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  2bao.cif.gz 2bao.cif.gz | 46.9 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb2bao.ent.gz pdb2bao.ent.gz | 31.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  2bao.json.gz 2bao.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  2bao_validation.pdf.gz 2bao_validation.pdf.gz | 359.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  2bao_full_validation.pdf.gz 2bao_full_validation.pdf.gz | 426 KB | 表示 | |
| XML形式データ |  2bao_validation.xml.gz 2bao_validation.xml.gz | 13.5 KB | 表示 | |
| CIF形式データ |  2bao_validation.cif.gz 2bao_validation.cif.gz | 17.2 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/ba/2bao https://data.pdbj.org/pub/pdb/validation_reports/ba/2bao ftp://data.pdbj.org/pub/pdb/validation_reports/ba/2bao ftp://data.pdbj.org/pub/pdb/validation_reports/ba/2bao | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 1064.279 Da / 分子数: 1 / 断片: N-terminal peptide / 由来タイプ: 合成 詳細: This sequence is the N-terminal peptide of a protein which occurs naturally in Homo sapiens (human). 参照: UniProt: P62330 |
|---|---|
| #2: 化合物 | ChemComp-MYR / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||
| NMR実験の詳細 | Text: This structure was determined using standard 2D homonuclear techniques |
- 試料調製
試料調製
| 詳細 | 内容: 1mM Myristoylated N-terminal Arf6; 5mM acetate buffer 溶媒系: 90% H2O/10% D2O |
|---|---|
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M |
|---|---|
| 放射波長 | 相対比: 1 |
| NMRスペクトロメーター | タイプ: Varian INOVA / 製造業者: Varian / モデル: INOVA / 磁場強度: 500 MHz |
- 解析
解析
| NMR software | 名称:  X-PLOR / バージョン: NIH-2.9.9 / 開発者: Lakowski / 分類: 精密化 X-PLOR / バージョン: NIH-2.9.9 / 開発者: Lakowski / 分類: 精密化 |
|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 |
| 代表構造 | 選択基準: lowest energy |
| NMRアンサンブル | コンフォーマー選択の基準: structures with the lowest energy 計算したコンフォーマーの数: 50 / 登録したコンフォーマーの数: 12 |
 ムービー
ムービー コントローラー
コントローラー





 PDBj
PDBj










