| 登録情報 | データベース: PDB / ID: 1ybl
|
|---|
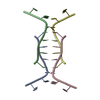
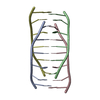
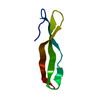
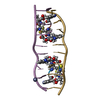
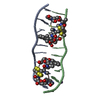
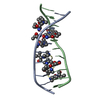
| タイトル | NMR structure of the i-motif tetramer of d(AACCCC) |
|---|
 要素 要素 | 5'-D(*AP*AP*CP*CP*CP*C)-3'  キーワード キーワード | DNA / i-motif / hemiprotonated cytidine / tetrahymena / telomer |
|---|
| 機能・相同性 | DNA 機能・相同性情報 機能・相同性情報 |
|---|
| 生物種 | synthetic construct (人工物) |
|---|
| 手法 | 溶液NMR / simulated annealing |
|---|
 データ登録者 データ登録者 | Esmaili, N. / Leroy, J.-L. |
|---|
 引用 引用 |  ジャーナル: Nucleic Acids Res. / 年: 2005 ジャーナル: Nucleic Acids Res. / 年: 2005
タイトル: i-motif solution structure and dynamics of the d(AACCCC) and d(CCCCAA) tetrahymena telomeric repeats
著者: Esmaili, N. / Leroy, J.-L. |
|---|
| 履歴 | | 登録 | 2004年12月21日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2005年1月11日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月30日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2018年6月6日 | Group: Data collection / Derived calculations ...Data collection / Derived calculations / Experimental preparation / Source and taxonomy
カテゴリ: ndb_struct_conf_na / ndb_struct_na_base_pair ...ndb_struct_conf_na / ndb_struct_na_base_pair / ndb_struct_na_base_pair_step / pdbx_entity_src_syn / pdbx_nmr_exptl_sample_conditions / pdbx_struct_assembly / pdbx_struct_assembly_prop / pdbx_struct_oper_list / struct_conn
Item: _ndb_struct_na_base_pair.hbond_type_28 / _ndb_struct_na_base_pair_step.tip ..._ndb_struct_na_base_pair.hbond_type_28 / _ndb_struct_na_base_pair_step.tip / _ndb_struct_na_base_pair_step.twist / _pdbx_nmr_exptl_sample_conditions.temperature |
|---|
| 改定 1.4 | 2024年5月1日 | Group: Data collection / Database references / カテゴリ: chem_comp_atom / chem_comp_bond / database_2
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession |
|---|
|
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 データ登録者
データ登録者 引用
引用 ジャーナル: Nucleic Acids Res. / 年: 2005
ジャーナル: Nucleic Acids Res. / 年: 2005 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1ybl.cif.gz
1ybl.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1ybl.ent.gz
pdb1ybl.ent.gz PDB形式
PDB形式 1ybl.json.gz
1ybl.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1ybl_validation.pdf.gz
1ybl_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1ybl_full_validation.pdf.gz
1ybl_full_validation.pdf.gz 1ybl_validation.xml.gz
1ybl_validation.xml.gz 1ybl_validation.cif.gz
1ybl_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/yb/1ybl
https://data.pdbj.org/pub/pdb/validation_reports/yb/1ybl ftp://data.pdbj.org/pub/pdb/validation_reports/yb/1ybl
ftp://data.pdbj.org/pub/pdb/validation_reports/yb/1ybl リンク
リンク 集合体
集合体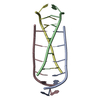
 要素
要素 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー







 PDBj
PDBj

 X-PLOR
X-PLOR