| 登録情報 | データベース: PDB / ID: 1xtn
|
|---|
| タイトル | crystal structure of CISK-PX domain with sulfates |
|---|
 要素 要素 | Serine/threonine-protein kinase Sgk3 |
|---|
 キーワード キーワード | TRANSFERASE / CISK / PX domain |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Stimuli-sensing channels / negative regulation of extrinsic apoptotic signaling pathway in absence of ligand / potassium channel regulator activity / localization / phosphatidylinositol binding / recycling endosome / cytoplasmic vesicle / molecular adaptor activity / early endosome / non-specific serine/threonine protein kinase ...Stimuli-sensing channels / negative regulation of extrinsic apoptotic signaling pathway in absence of ligand / potassium channel regulator activity / localization / phosphatidylinositol binding / recycling endosome / cytoplasmic vesicle / molecular adaptor activity / early endosome / non-specific serine/threonine protein kinase / intracellular signal transduction / phosphorylation / protein serine kinase activity / protein serine/threonine kinase activity / ATP binding / nucleus類似検索 - 分子機能 Serine/threonine-protein kinase Sgk3, catalytic domain / CISK, PX domain / Phox-like domain / PX Domain / PhoX homologous domain, present in p47phox and p40phox. / PX domain profile. / PX domain / Phox homology / PX domain superfamily / Protein kinase, C-terminal ...Serine/threonine-protein kinase Sgk3, catalytic domain / CISK, PX domain / Phox-like domain / PX Domain / PhoX homologous domain, present in p47phox and p40phox. / PX domain profile. / PX domain / Phox homology / PX domain superfamily / Protein kinase, C-terminal / Protein kinase C terminal domain / Extension to Ser/Thr-type protein kinases / AGC-kinase, C-terminal / AGC-kinase C-terminal domain profile. / Serine/threonine-protein kinase, active site / Serine/Threonine protein kinases active-site signature. / Protein kinase domain / Serine/Threonine protein kinases, catalytic domain / Protein kinase, ATP binding site / Protein kinases ATP-binding region signature. / Protein kinase domain profile. / Protein kinase domain / Protein kinase-like domain superfamily / 2-Layer Sandwich / Alpha Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Mus musculus (ハツカネズミ) Mus musculus (ハツカネズミ) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.2 Å 分子置換 / 解像度: 2.2 Å |
|---|
 データ登録者 データ登録者 | Xing, Y. / Liu, D. / Zhang, R. / Joachimiak, A. / Songyang, Z. / Xu, W. |
|---|
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2004 ジャーナル: J.Biol.Chem. / 年: 2004
タイトル: Structural basis of membrane targeting by the Phox homology domain of cytokine-independent survival kinase (CISK-PX)
著者: Xing, Y. / Liu, D. / Zhang, R. / Joachimiak, A. / Songyang, Z. / Xu, W. |
|---|
| 履歴 | | 登録 | 2004年10月22日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2004年11月2日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月30日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2018年1月24日 | Group: Database references / Structure summary / カテゴリ: audit_author / citation_author / Item: _audit_author.name / _citation_author.name |
|---|
| 改定 1.4 | 2023年8月23日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Refinement description
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_initial_refinement_model / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 2.2 Å
分子置換 / 解像度: 2.2 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2004
ジャーナル: J.Biol.Chem. / 年: 2004 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1xtn.cif.gz
1xtn.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1xtn.ent.gz
pdb1xtn.ent.gz PDB形式
PDB形式 1xtn.json.gz
1xtn.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1xtn_validation.pdf.gz
1xtn_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1xtn_full_validation.pdf.gz
1xtn_full_validation.pdf.gz 1xtn_validation.xml.gz
1xtn_validation.xml.gz 1xtn_validation.cif.gz
1xtn_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/xt/1xtn
https://data.pdbj.org/pub/pdb/validation_reports/xt/1xtn ftp://data.pdbj.org/pub/pdb/validation_reports/xt/1xtn
ftp://data.pdbj.org/pub/pdb/validation_reports/xt/1xtn リンク
リンク 集合体
集合体
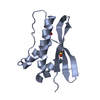

 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  ALS
ALS  / ビームライン: 5.0.1 / 波長: 1 Å
/ ビームライン: 5.0.1 / 波長: 1 Å 解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj





