+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1xfw | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of anthrax edema factor (EF) in complex with calmodulin and 3'5' cyclic AMP (cAMP) | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | LYASE/METAL BINDING PROTEIN / protein-protein interaction / LYASE-METAL BINDING PROTEIN COMPLEX | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報symbiont-mediated perturbation of host signal transduction pathway / symbiont-mediated cAMP intoxication of host cell / calcium- and calmodulin-responsive adenylate cyclase activity / : / : / positive regulation of cyclic-nucleotide phosphodiesterase activity / : / adenylate cyclase / negative regulation of peptidyl-threonine phosphorylation / cAMP biosynthetic process ...symbiont-mediated perturbation of host signal transduction pathway / symbiont-mediated cAMP intoxication of host cell / calcium- and calmodulin-responsive adenylate cyclase activity / : / : / positive regulation of cyclic-nucleotide phosphodiesterase activity / : / adenylate cyclase / negative regulation of peptidyl-threonine phosphorylation / cAMP biosynthetic process / establishment of protein localization to mitochondrial membrane / type 3 metabotropic glutamate receptor binding / adenylate cyclase activity / CaM pathway / Cam-PDE 1 activation / Sodium/Calcium exchangers / Calmodulin induced events / positive regulation of peptidyl-threonine phosphorylation / positive regulation of ryanodine-sensitive calcium-release channel activity / Reduction of cytosolic Ca++ levels / Activation of Ca-permeable Kainate Receptor / CREB1 phosphorylation through the activation of CaMKII/CaMKK/CaMKIV cascasde / host cell cytosol / positive regulation of DNA binding / Loss of phosphorylation of MECP2 at T308 / CREB1 phosphorylation through the activation of Adenylate Cyclase / CaMK IV-mediated phosphorylation of CREB / PKA activation / negative regulation of high voltage-gated calcium channel activity / Glycogen breakdown (glycogenolysis) / CLEC7A (Dectin-1) induces NFAT activation / response to corticosterone / Activation of RAC1 downstream of NMDARs / organelle localization by membrane tethering / negative regulation of ryanodine-sensitive calcium-release channel activity / mitochondrion-endoplasmic reticulum membrane tethering / autophagosome membrane docking / negative regulation of calcium ion export across plasma membrane / regulation of cardiac muscle cell action potential / presynaptic endocytosis / nitric-oxide synthase binding / small molecule binding / Synthesis of IP3 and IP4 in the cytosol / regulation of cell communication by electrical coupling involved in cardiac conduction / Phase 0 - rapid depolarisation / regulation of synaptic vesicle exocytosis / calcineurin-mediated signaling / Negative regulation of NMDA receptor-mediated neuronal transmission / Unblocking of NMDA receptors, glutamate binding and activation / positive regulation of protein autophosphorylation / RHO GTPases activate PAKs / Ion transport by P-type ATPases / Uptake and function of anthrax toxins / regulation of ryanodine-sensitive calcium-release channel activity / Long-term potentiation / protein phosphatase activator activity / Calcineurin activates NFAT / adenylate cyclase binding / Regulation of MECP2 expression and activity / DARPP-32 events / catalytic complex / Smooth Muscle Contraction / regulation of synaptic vesicle endocytosis / detection of calcium ion / regulation of cardiac muscle contraction / positive regulation of protein serine/threonine kinase activity / RHO GTPases activate IQGAPs / regulation of cardiac muscle contraction by regulation of the release of sequestered calcium ion / calcium channel inhibitor activity / activation of adenylate cyclase activity / cellular response to interferon-beta / Protein methylation / presynaptic cytosol / phosphatidylinositol 3-kinase binding / Activation of AMPK downstream of NMDARs / Ion homeostasis / regulation of release of sequestered calcium ion into cytosol by sarcoplasmic reticulum / eNOS activation / positive regulation of nitric-oxide synthase activity / titin binding / enzyme regulator activity / Tetrahydrobiopterin (BH4) synthesis, recycling, salvage and regulation / sperm midpiece / voltage-gated potassium channel complex / regulation of calcium-mediated signaling / calcium channel complex / substantia nigra development / FCERI mediated Ca+2 mobilization / response to amphetamine / Ras activation upon Ca2+ influx through NMDA receptor / regulation of heart rate / FCGR3A-mediated IL10 synthesis / calyx of Held / nitric-oxide synthase regulator activity / Antigen activates B Cell Receptor (BCR) leading to generation of second messengers / adenylate cyclase activator activity / sarcomere / VEGFR2 mediated cell proliferation / regulation of cytokinesis / protein serine/threonine kinase activator activity 類似検索 - 分子機能 | ||||||
| 生物種 |   Homo sapiens (ヒト) Homo sapiens (ヒト) | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 3.4 Å 分子置換 / 解像度: 3.4 Å | ||||||
 データ登録者 データ登録者 | Shen, Y. / Zhukovskaya, N.L. / Guo, Q. / Florian, J. / Tang, W.J. | ||||||
 引用 引用 |  ジャーナル: EMBO J. / 年: 2005 ジャーナル: EMBO J. / 年: 2005タイトル: Calcium-independent calmodulin binding and two-metal-ion catalytic mechanism of anthrax edema factor. 著者: Shen, Y. / Zhukovskaya, N.L. / Guo, Q. / Florian, J. / Tang, W.J. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1xfw.cif.gz 1xfw.cif.gz | 1 MB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1xfw.ent.gz pdb1xfw.ent.gz | 862.4 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1xfw.json.gz 1xfw.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1xfw_validation.pdf.gz 1xfw_validation.pdf.gz | 2.4 MB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1xfw_full_validation.pdf.gz 1xfw_full_validation.pdf.gz | 2.9 MB | 表示 | |
| XML形式データ |  1xfw_validation.xml.gz 1xfw_validation.xml.gz | 246.2 KB | 表示 | |
| CIF形式データ |  1xfw_validation.cif.gz 1xfw_validation.cif.gz | 319.9 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/xf/1xfw https://data.pdbj.org/pub/pdb/validation_reports/xf/1xfw ftp://data.pdbj.org/pub/pdb/validation_reports/xf/1xfw ftp://data.pdbj.org/pub/pdb/validation_reports/xf/1xfw | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 3 | 
| ||||||||
| 4 | 
| ||||||||
| 5 | 
| ||||||||
| 6 | 
| ||||||||
| 7 | 
| ||||||||
| 8 | 
| ||||||||
| 9 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
-タンパク質 , 2種, 12分子 ABCDEFOPQRST
| #1: タンパク質 | 分子量: 90128.898 Da / 分子数: 6 / 由来タイプ: 組換発現 / 由来: (組換発現)   #2: タンパク質 | 分子量: 16851.561 Da / 分子数: 6 / 由来タイプ: 組換発現 / 由来: (組換発現)  Homo sapiens (ヒト) / 発現宿主: Homo sapiens (ヒト) / 発現宿主:  |
|---|
-非ポリマー , 4種, 36分子 
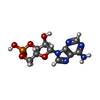





| #3: 化合物 | ChemComp-MG / #4: 化合物 | ChemComp-CMP / #5: 化合物 | ChemComp-CA / #6: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.1 Å3/Da / 溶媒含有率: 60 % |
|---|---|
| 結晶化 | 温度: 278 K / 手法: 蒸気拡散法, ハンギングドロップ法 / pH: 8.5 詳細: PEG400, magnesium chloride, pH 8.5, VAPOR DIFFUSION, HANGING DROP, temperature 278K |
-データ収集
| 回折 | 平均測定温度: 190 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  APS APS  / ビームライン: 14-ID-B / 波長: 0.98 Å / ビームライン: 14-ID-B / 波長: 0.98 Å |
| 検出器 | タイプ: MACSCIENCE / 検出器: IMAGE PLATE / 日付: 2003年11月28日 |
| 放射 | モノクロメーター: graphite / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 0.98 Å / 相対比: 1 |
| 反射 | 解像度: 3.3→100 Å / Num. obs: 106659 / % possible obs: 99 % / Observed criterion σ(F): 2 / Observed criterion σ(I): 3 / 冗長度: 4.2 % / Rmerge(I) obs: 0.094 / Rsym value: 0.067 / Net I/σ(I): 14 |
| 反射 シェル | 解像度: 3.3→3.42 Å / 冗長度: 3.7 % / Rmerge(I) obs: 0.555 / Mean I/σ(I) obs: 2.4 / Num. unique all: 120342 / Rsym value: 0.465 / % possible all: 100 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 / 解像度: 3.4→29.68 Å / Rfactor Rfree error: 0.004 / Data cutoff high absF: 517178.02 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber 分子置換 / 解像度: 3.4→29.68 Å / Rfactor Rfree error: 0.004 / Data cutoff high absF: 517178.02 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / 交差検証法: THROUGHOUT / σ(F): 0 / 立体化学のターゲット値: Engh & Huber
| ||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: FLAT MODEL / Bsol: 48.2411 Å2 / ksol: 0.26171 e/Å3 | ||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 74.9 Å2
| ||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 3.4→29.68 Å
| ||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: CONSTR | ||||||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 3.4→3.61 Å / Rfactor Rfree error: 0.013 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||
| Xplor file |
|
 ムービー
ムービー コントローラー
コントローラー























 PDBj
PDBj



























