+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1vm4 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Solution structure of an antibacterial and antitumor peptide designed based on the N-terminal membrane anchor of E. coli enzyme IIA (Glucose) | ||||||
 要素 要素 | peptide A4 | ||||||
 キーワード キーワード | ANTIBIOTIC / amphipathic helix / antimicrobial peptide / bacterial membrane anchor / anticancer peptide | ||||||
| 生物種 | synthetic construct (人工物) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | Wang, G. / Li, X. | ||||||
 引用 引用 |  ジャーナル: J.Biol.Chem. / 年: 2005 ジャーナル: J.Biol.Chem. / 年: 2005タイトル: Correlation of Three-dimensional Structures with the Antibacterial Activity of a Group of Peptides Designed Based on a Nontoxic Bacterial Membrane Anchor. 著者: Wang, G. / Li, Y. / Li, X. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1vm4.cif.gz 1vm4.cif.gz | 26.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1vm4.ent.gz pdb1vm4.ent.gz | 17.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1vm4.json.gz 1vm4.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1vm4_validation.pdf.gz 1vm4_validation.pdf.gz | 342.9 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1vm4_full_validation.pdf.gz 1vm4_full_validation.pdf.gz | 350.8 KB | 表示 | |
| XML形式データ |  1vm4_validation.xml.gz 1vm4_validation.xml.gz | 3.3 KB | 表示 | |
| CIF形式データ |  1vm4_validation.cif.gz 1vm4_validation.cif.gz | 4 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/vm/1vm4 https://data.pdbj.org/pub/pdb/validation_reports/vm/1vm4 ftp://data.pdbj.org/pub/pdb/validation_reports/vm/1vm4 ftp://data.pdbj.org/pub/pdb/validation_reports/vm/1vm4 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 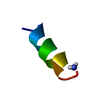
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 1480.770 Da / 分子数: 1 / 由来タイプ: 合成 詳細: The peptide was synthesized using the solid-phase method and purified by HPLC. The template sequence of the peptide is naturally found in Escherichia coli (bacteria). 由来: (合成) synthetic construct (人工物) |
|---|---|
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||||||||||||||
| NMR実験の詳細 | Text: This structure was determined using standard 2D homonuclear NMR techniques, plus the use of backbone angle restraints derived from a set of heteronuclear chemical shifts measured on the natural ...Text: This structure was determined using standard 2D homonuclear NMR techniques, plus the use of backbone angle restraints derived from a set of heteronuclear chemical shifts measured on the natural abundance peptide bound to micelles. |
- 試料調製
試料調製
| 詳細 | 内容: 2mM peptide, 80mM sodium dodecylsulfate, 90% H2O and 10% D2O 溶媒系: 90% H2O and 10% D2O |
|---|---|
| 試料状態 | pH: 5.4 / 圧: ambient / 温度: 298 K |
-NMR測定
| NMRスペクトロメーター | タイプ: Varian INOVA / 製造業者: Varian / モデル: INOVA / 磁場強度: 600 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: The structures are based on 109 distances derived from the NOESY spectra, 22 backbone dihedral angles derived from a set of chemical shifts using the NMR program TALOS, and 5 chi1 angle restraints. | ||||||||||||||||||||
| 代表構造 | 選択基準: most resemble the average structure | ||||||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: No NOE violations greater than 0.50, rms difference for bond deviations from ideality less than 0.01 A, rms difference for angle deviations from ideality less ...コンフォーマー選択の基準: No NOE violations greater than 0.50, rms difference for bond deviations from ideality less than 0.01 A, rms difference for angle deviations from ideality less than 5 degrees, Structures with the lowerest energies in the ensemble. 計算したコンフォーマーの数: 100 / 登録したコンフォーマーの数: 5 |
 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj HSQC
HSQC