登録構造単位
A: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
B: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
C: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
D: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
E: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
F: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細
分子量 (理論値) 分子数 合計 (水以外) 100,985 37 ポリマ- 97,019 6 非ポリマー 3,966 31 水 12,791 710
1
A: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
A: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
A: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,716 21 ポリマ- 48,510 3 非ポリマー 2,206 18 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_655 -y+1,x-y,z 1 crystal symmetry operation 3_665 -x+y+1,-x+1,z 1
2
B: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
B: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
B: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,428 18 ポリマ- 48,510 3 非ポリマー 1,918 15 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_665 -y+1,x-y+1,z 1 crystal symmetry operation 3_565 -x+y,-x+1,z 1
3
C: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
C: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
C: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,428 18 ポリマ- 48,510 3 非ポリマー 1,918 15 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_555 -y,x-y,z 1 crystal symmetry operation 3_555 -x+y,-x,z 1
4
D: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
D: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
D: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,530 18 ポリマ- 48,510 3 非ポリマー 2,020 15 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 3_665 -x+y+1,-x+1,z 1 crystal symmetry operation 2_655 -y+1,x-y,z 1
5
E: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
E: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
E: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,428 18 ポリマ- 48,510 3 非ポリマー 1,918 15 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_645 -y+1,x-y-1,z 1 crystal symmetry operation 3_765 -x+y+2,-x+1,z 1
6
F: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
F: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子
F: DR HEMAGGLUTININ STRUCTURAL SUBUNIT
ヘテロ分子 概要 構成要素の詳細 対称操作 計算値
分子量 (理論値) 分子数 合計 (水以外) 50,428 18 ポリマ- 48,510 3 非ポリマー 1,918 15 水 54 3
タイプ 名称 対称操作 数 identity operation 1_555 x,y,z 1 crystal symmetry operation 2_755 -y+2,x-y,z 1 crystal symmetry operation 3_775 -x+y+2,-x+2,z 1
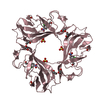
単位格子 Length a, b, c (Å) 118.969, 118.969, 57.416 Angle α, β, γ (deg.) 90.00, 90.00, 120.00 Int Tables number 143 Space group name H-M P3
非結晶学的対称性 (NCS) NCS oper ID Code Matrix ベクター 1 given(-0.50111, 0.86537, -0.00541), (-0.86538, -0.5011, 0.00269), (-0.00039, 0.00603, 0.99998)0.06384, 68.71037, -3.679052 given(-0.37085, -0.92862, -0.01147), (0.92864, -0.37093, 0.00595), (-0.00978, -0.00845, 0.99992)59.50617, 34.36259, -1.051853 given(-0.99745, 0.07115, -0.00525), (0.07111, 0.99744, 0.00812), (0.00581, 0.00773, -0.99995)116.5445, -4.5186, 29.284184 given(0.55986, -0.82858, -0.00128), (-0.82858, -0.55987, 0.00236), (-0.00267, -0.00026, -1)-7.10494, 132.77824, 31.451495 given(0.55521, 0.83167, -0.00822), (0.8317, -0.55513, 0.01046), (0.00414, -0.01265, -0.99991)-63.35427, -26.87984, 33.52893
詳細 THERE ARE SIX TRIMERS IN THE CRYSTAL CELL , EACH LINKEDINTERNALLY BY 3 INTERMOLECULAR DISULPHIDE BONDS OF THEMONOMER WITH TWO OF ITS THREEFOLD-RELATED COPIES
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.9 Å
分子置換 / 解像度: 1.9 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Biol.Chem. / 年: 2004
ジャーナル: J.Biol.Chem. / 年: 2004 ジャーナル: Mol.Cell / 年: 2004
ジャーナル: Mol.Cell / 年: 2004 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1usq.cif.gz
1usq.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1usq.ent.gz
pdb1usq.ent.gz PDB形式
PDB形式 1usq.json.gz
1usq.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/us/1usq
https://data.pdbj.org/pub/pdb/validation_reports/us/1usq ftp://data.pdbj.org/pub/pdb/validation_reports/us/1usq
ftp://data.pdbj.org/pub/pdb/validation_reports/us/1usq リンク
リンク 集合体
集合体





 要素
要素

 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  SRS
SRS  / ビームライン: PX9.6 / 波長: 0.87
/ ビームライン: PX9.6 / 波長: 0.87  解析
解析 分子置換
分子置換 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj