+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1rms | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | CRYSTAL STRUCTURES OF RIBONUCLEASE MS COMPLEXED WITH 3'-GUANYLIC ACID A GP*C ANALOGUE, 2'-DEOXY-2'-FLUOROGUANYLYL-3',5'-CYTIDINE | ||||||
 要素 要素 | RIBONUCLEASE MS | ||||||
 キーワード キーワード | HYDROLASE(ENDORIBONUCLEASE) | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報ribonuclease T1 / ribonuclease T1 activity / RNA endonuclease activity / lyase activity / RNA binding 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / 解像度: 1.9 Å X線回折 / 解像度: 1.9 Å | ||||||
 データ登録者 データ登録者 | Nonaka, T. / Mitsui, Y. / Nakamura, K.T. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1993 ジャーナル: Biochemistry / 年: 1993タイトル: Crystal structure of ribonuclease Ms (as a ribonuclease T1 homologue) complexed with a guanylyl-3',5'-cytidine analogue. 著者: Nonaka, T. / Nakamura, K.T. / Uesugi, S. / Ikehara, M. / Irie, M. / Mitsui, Y. #1:  ジャーナル: FEBS Lett. / 年: 1991 ジャーナル: FEBS Lett. / 年: 1991タイトル: Three-Dimensional Structure of Ribonuclease Ms(Asterisk)3'-Guanylic Acid Complex at 2.5A Resolution 著者: Nonaka, T. / Mitsui, Y. / Irie, M. / Nakamura, K.T. #2:  ジャーナル: J.Mol.Biol. / 年: 1989 ジャーナル: J.Mol.Biol. / 年: 1989タイトル: Crystallization of a Complex between Ribonuclease Ms and 3'-Guanylic Acid 著者: Mitsui, T.Nonaka Y. / Nakamura, K.T. / Watanabe, H. / Ohgi, K. / Irie, M. #3:  ジャーナル: J.Biol.Chem. / 年: 1988 ジャーナル: J.Biol.Chem. / 年: 1988タイトル: Three-Dimensional Structure of the Ribonuclease T1(Asterisk)2'-Gmp Complex at 1.9-A Resolution 著者: Arni, R. / Heinemann, U. / Tokuoka, R. / Saenger, W. | ||||||
| 履歴 |
| ||||||
| Remark 700 | SHEET ASP 83 (POSITION 1), GLU 84 (POSITION 2) AND ASN 80 (POSITION X) FORM A "G1 TYPE" BETA BULGE. ...SHEET ASP 83 (POSITION 1), GLU 84 (POSITION 2) AND ASN 80 (POSITION X) FORM A "G1 TYPE" BETA BULGE. GLU 84 AND ASN 80 ALSO OCCUPY RESPECTIVELY POSITIONS 0 AND X-1 OF A "WIDE TYPE" BETA BULGE (SEE REMARK 9). ALA 86 (POSITION 1), GLY 87 (POSITION 2) AND ILE 78 (POSITION X) FORM A "CLASSIC TYPE" BETA BULGE. ALA 86 AND ILE 78 ALSO OCCUPY POSITIONS 3 AND X+1 OF A "WIDE TYPE" BETA BULGE (SEE REMARK 9). GLU 4 (POSITION 1), TYR 5 (POSITION 2) AND TYR 12 (POSITION X) FORM A "CLASSIC TYPE" BETA BULGE. GLU 84 (POSITION 0), LEU 85 (POSITION 1) ALA 86 (POSITION 3) ILE 78 (POSITION X-1), PHE 79 (POSITION X) AND ASN 80 (POSITION X+1) FORM A "WIDE TYPE" BETA BULGE (SEE REMARKS 6 AND 7). |
- 構造の表示
構造の表示
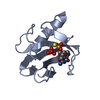
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1rms.cif.gz 1rms.cif.gz | 34.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1rms.ent.gz pdb1rms.ent.gz | 22.9 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1rms.json.gz 1rms.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1rms_validation.pdf.gz 1rms_validation.pdf.gz | 457.7 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1rms_full_validation.pdf.gz 1rms_full_validation.pdf.gz | 464.4 KB | 表示 | |
| XML形式データ |  1rms_validation.xml.gz 1rms_validation.xml.gz | 4.7 KB | 表示 | |
| CIF形式データ |  1rms_validation.cif.gz 1rms_validation.cif.gz | 6.5 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/rm/1rms https://data.pdbj.org/pub/pdb/validation_reports/rm/1rms ftp://data.pdbj.org/pub/pdb/validation_reports/rm/1rms ftp://data.pdbj.org/pub/pdb/validation_reports/rm/1rms | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| Atom site foot note | 1: RESIDUE 38 IS A CIS PROLINE. 2: THE PEPTIDE LINKAGE GLY 53-THR 54 HAS BEEN FOUND TO BE IN THE CIS-CONFORMATION AFTER LEAST-SQUARES REFINEMENT. |
- 要素
要素
| #1: タンパク質 | 分子量: 11409.980 Da / 分子数: 1 / 由来タイプ: 組換発現 / 由来: (組換発現)  |
|---|---|
| #2: 化合物 | ChemComp-3GP / |
| #3: 水 | ChemComp-HOH / |
| Has protein modification | Y |
-実験情報
-実験
| 実験 | 手法:  X線回折 X線回折 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.45 Å3/Da / 溶媒含有率: 49.9 % |
|---|
-データ収集
| 放射 | 散乱光タイプ: x-ray |
|---|---|
| 放射波長 | 相対比: 1 |
| 反射 | *PLUS 最高解像度: 1.9 Å / Num. obs: 9223 / Num. measured all: 10035 / Rmerge(I) obs: 0.116 |
- 解析
解析
| ソフトウェア | 名称: PROLSQ / 分類: 精密化 | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 解像度: 1.9→6 Å 詳細: THE RMS DEVIATION BETWEEN THE SUPERIMPOSED CA POSITIONS OF RNASE MS AND RNASE T1 (REFERENCE 3, PROTEIN DATA BANK ENTRY 1RNT) EXCLUDING THE FOUR N-TERMINAL RESIDUES IS 0.55 ANGSTROMS. ...詳細: THE RMS DEVIATION BETWEEN THE SUPERIMPOSED CA POSITIONS OF RNASE MS AND RNASE T1 (REFERENCE 3, PROTEIN DATA BANK ENTRY 1RNT) EXCLUDING THE FOUR N-TERMINAL RESIDUES IS 0.55 ANGSTROMS. SIGNIFICANT DEVIATION OCCURS ONLY AT THE N-TERMINUS EXCEPT FOR A LOOP-OUT PORTION WHICH INDICATES THAT THE T1 SEQUENCE, GLY 34-SER 35-ASN 36-SER 37, SHOULD BE ALIGNED WITH THE MS SEQUENCE ASP-35-GAP-GAP-ASP 36. THE FOLLOWING CRITERIA WERE ADOPTED FOR ASSIGNING MAIN-CHAIN HYDROGEN BONDS: THE MAXIMUM DISTANCE BETWEEN AN AMIDE HYDROGEN AND A CARBONYL OXYGEN IS 2.60 ANGSTROMS. THE MAXIMUM DISTANCE BETWEEN AN AMIDE NITROGEN AND A CARBONYL OXYGEN IS 3.35.ANGSTROMS. THE MINIMUM NHO ANGLE IS 100 DEGREES. THE MINIMUM COH ANGLE IS 100 DEGREES. THE MINIMUM CON ANGLE IS 100 DEGREES. HYDROGEN ATOM POSITIONS WERE CALCULATED USING THE PROGRAM *X-PLOR*. THE ASSIGNMENT OF BETA-TURN TYPES IS BASED ON WILMOT AND THORNTON (C.M.WILMOT AND J.M.THORNTON, PROTEIN ENGINEERING, 3, 479-493, 1990). PHI,PSI ANGLES ARE ALLOWED TO DEVIATE BY 30 DEGREES FROM THEIR IDEAL VALUES.
| ||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.9→6 Å
| ||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー













 PDBj
PDBj