| 登録情報 | データベース: PDB / ID: 1rgk
|
|---|
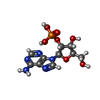
| タイトル | RNASE T1 MUTANT GLU46GLN BINDS THE INHIBITORS 2'GMP AND 2'AMP AT THE 3' SUBSITE |
|---|
 要素 要素 | RIBONUCLEASE T1 |
|---|
 キーワード キーワード | HYDROLASE(ENDORIBONUCLEASE) |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
hyphal tip / ribonuclease T1 / ribonuclease T1 activity / cell septum / RNA endonuclease activity / lyase activity / RNA binding類似検索 - 分子機能 : / : / Microbial ribonucleases / Guanine-specific ribonuclease N1/T1/U2 / Ribonuclease/ribotoxin / ribonuclease / Nuclear Transport Factor 2; Chain: A, / Roll / Alpha Beta類似検索 - ドメイン・相同性 ADENOSINE-2'-MONOPHOSPHATE / Guanyl-specific ribonuclease T1類似検索 - 構成要素 |
|---|
| 生物種 |   Aspergillus oryzae (米麹菌) Aspergillus oryzae (米麹菌) |
|---|
| 手法 |  X線回折 / 解像度: 1.87 Å X線回折 / 解像度: 1.87 Å |
|---|
 データ登録者 データ登録者 | Granzin, J. / Puras-Lutzke, R. / Landt, O. / Grunert, H.-P. / Heinemann, U. / Saenger, W. / Hahn, U. |
|---|
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 1992 ジャーナル: J.Mol.Biol. / 年: 1992
タイトル: RNase T1 mutant Glu46Gln binds the inhibitors 2'GMP and 2'AMP at the 3' subsite.
著者: Granzin, J. / Puras-Lutzke, R. / Landt, O. / Grunert, H.P. / Heinemann, U. / Saenger, W. / Hahn, U. |
|---|
| 履歴 | | 登録 | 1992年2月19日 | 処理サイト: BNL |
|---|
| 改定 1.0 | 1993年1月15日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年3月24日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2017年11月29日 | Group: Derived calculations / Other
カテゴリ: pdbx_database_status / struct_conf / struct_conf_type
Item: _pdbx_database_status.process_site |
|---|
| 改定 1.4 | 2024年10月23日 | Group: Data collection / Database references ...Data collection / Database references / Derived calculations / Structure summary
カテゴリ: chem_comp_atom / chem_comp_bond ...chem_comp_atom / chem_comp_bond / database_2 / pdbx_entry_details / pdbx_modification_feature / pdbx_struct_conn_angle / struct_conn / struct_ref_seq_dif / struct_site
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_struct_conn_angle.ptnr1_auth_comp_id / _pdbx_struct_conn_angle.ptnr1_auth_seq_id / _pdbx_struct_conn_angle.ptnr1_label_asym_id / _pdbx_struct_conn_angle.ptnr1_label_atom_id / _pdbx_struct_conn_angle.ptnr1_label_comp_id / _pdbx_struct_conn_angle.ptnr1_label_seq_id / _pdbx_struct_conn_angle.ptnr3_auth_comp_id / _pdbx_struct_conn_angle.ptnr3_auth_seq_id / _pdbx_struct_conn_angle.ptnr3_label_asym_id / _pdbx_struct_conn_angle.ptnr3_label_atom_id / _pdbx_struct_conn_angle.ptnr3_label_comp_id / _pdbx_struct_conn_angle.ptnr3_label_seq_id / _pdbx_struct_conn_angle.value / _struct_conn.pdbx_dist_value / _struct_conn.ptnr1_auth_comp_id / _struct_conn.ptnr1_auth_seq_id / _struct_conn.ptnr1_label_asym_id / _struct_conn.ptnr1_label_atom_id / _struct_conn.ptnr1_label_comp_id / _struct_conn.ptnr1_label_seq_id / _struct_conn.ptnr2_auth_comp_id / _struct_conn.ptnr2_auth_seq_id / _struct_conn.ptnr2_label_asym_id / _struct_conn.ptnr2_label_atom_id / _struct_conn.ptnr2_label_comp_id / _struct_conn.ptnr2_label_seq_id / _struct_ref_seq_dif.details / _struct_site.pdbx_auth_asym_id / _struct_site.pdbx_auth_comp_id / _struct_site.pdbx_auth_seq_id |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 X線回折 / 解像度: 1.87 Å
X線回折 / 解像度: 1.87 Å  データ登録者
データ登録者 引用
引用 ジャーナル: J.Mol.Biol. / 年: 1992
ジャーナル: J.Mol.Biol. / 年: 1992 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1rgk.cif.gz
1rgk.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1rgk.ent.gz
pdb1rgk.ent.gz PDB形式
PDB形式 1rgk.json.gz
1rgk.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/rg/1rgk
https://data.pdbj.org/pub/pdb/validation_reports/rg/1rgk ftp://data.pdbj.org/pub/pdb/validation_reports/rg/1rgk
ftp://data.pdbj.org/pub/pdb/validation_reports/rg/1rgk リンク
リンク 集合体
集合体
 要素
要素

 X線回折
X線回折 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー












 PDBj
PDBj