[English] 日本語
 Yorodumi
Yorodumi- PDB-1qtq: GLUTAMINYL-TRNA SYNTHETASE COMPLEXED WITH TRNA AND AN AMINO ACID ... -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1qtq | ||||||
|---|---|---|---|---|---|---|---|
| Title | GLUTAMINYL-TRNA SYNTHETASE COMPLEXED WITH TRNA AND AN AMINO ACID ANALOG | ||||||
 Components Components |
| ||||||
 Keywords Keywords | LIGASE/RNA / TRNA SYNTHETASE / GLUTAMINE / TRNAGLN / E. COLI / COMPLEX / LIGASE-RNA COMPLEX | ||||||
| Function / homology |  Function and homology information Function and homology informationglutamine-tRNA ligase / glutamine-tRNA ligase activity / glutaminyl-tRNA aminoacylation / glutamyl-tRNA aminoacylation / ATP binding / cytosol Similarity search - Function | ||||||
| Biological species |  | ||||||
| Method |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 2.25 Å MOLECULAR REPLACEMENT / Resolution: 2.25 Å | ||||||
 Authors Authors | Rath, V.L. / Silvian, L.F. / Beijer, B. / Sproat, B.S. / Steitz, T.A. | ||||||
 Citation Citation |  Journal: Structure / Year: 1998 Journal: Structure / Year: 1998Title: How glutaminyl-tRNA synthetase selects glutamine. Authors: Rath, V.L. / Silvian, L.F. / Beijer, B. / Sproat, B.S. / Steitz, T.A. | ||||||
| History |
|
- Structure visualization
Structure visualization
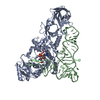
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1qtq.cif.gz 1qtq.cif.gz | 160.2 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1qtq.ent.gz pdb1qtq.ent.gz | 125.6 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1qtq.json.gz 1qtq.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/qt/1qtq https://data.pdbj.org/pub/pdb/validation_reports/qt/1qtq ftp://data.pdbj.org/pub/pdb/validation_reports/qt/1qtq ftp://data.pdbj.org/pub/pdb/validation_reports/qt/1qtq | HTTPS FTP |
|---|
-Related structure data
| Related structure data |  1gtrS S: Starting model for refinement |
|---|---|
| Similar structure data |
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| Unit cell |
|
- Components
Components
| #1: RNA chain | Mass: 24060.287 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   | ||||
|---|---|---|---|---|---|
| #2: Protein | Mass: 63434.641 Da / Num. of mol.: 1 Source method: isolated from a genetically manipulated source Source: (gene. exp.)   | ||||
| #3: Chemical | ChemComp-SO4 / #4: Chemical | ChemComp-QSI / | #5: Water | ChemComp-HOH / | |
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 3.67 Å3/Da / Density % sol: 70 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 290 K / Method: vapor diffusion, hanging drop / pH: 7 Details: 2.0 M AMMONIUM SULFATE, 20 MM MGSO4, 80 MM PIPES, PH 7.0, 1 MM QSI, AND 1:1 MOLAR RATIO OF TRNA TO PROTEIN AT 17 DEGREES C BY THE HANGING DROP METHOD., vapor diffusion - hanging drop, temperature 290.00K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Crystal | *PLUS Density % sol: 70 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Crystal grow | *PLUS Temperature: 17 ℃ / PH range low: 7.2 / PH range high: 6.8 | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 110 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  NSLS NSLS  / Beamline: X25 / Beamline: X25 |
| Detector | Type: MARRESEARCH / Detector: IMAGE PLATE / Date: May 1, 1995 / Details: PT COATED SI MIRROR |
| Radiation | Monochromator: SILICON / Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Relative weight: 1 |
| Reflection | Resolution: 2.25→28 Å / Num. obs: 51376 / % possible obs: 88.8 % / Observed criterion σ(I): 0 / Redundancy: 2.5 % / Biso Wilson estimate: 34.5 Å2 / Rmerge(I) obs: 0.084 |
| Reflection shell | Resolution: 2.25→2.3 Å / Redundancy: 1 % / Rmerge(I) obs: 0.534 / % possible all: 67.7 |
| Reflection | *PLUS Highest resolution: 2.25 Å / Lowest resolution: 28 Å / % possible obs: 88.8 % / Observed criterion σ(I): 0 / Redundancy: 2.5 % / Biso Wilson estimate: 34.5 Å2 |
| Reflection shell | *PLUS Highest resolution: 2.25 Å / Lowest resolution: 2.3 Å / % possible obs: 67.7 % / Redundancy: 1 % / Mean I/σ(I) obs: 2.9 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT MOLECULAR REPLACEMENTStarting model: PDB ENTRY 1GTR Resolution: 2.25→30 Å / Rfactor Rfree error: 0.004 / Data cutoff high absF: 1087461.2 / Data cutoff low absF: 0 / Isotropic thermal model: RESTRAINED / Cross valid method: THROUGHOUT / σ(F): 0 Details: BULK SOLVENT MODEL USED RESIDUES 1 - 7, 443 - 453, AND 548 - 553 OF THE PROTEIN AND NUCLEOTIDE 901 OF THE TRNA ARE DISORDERED IN THE CRYSTAL STRUCTURE.
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | Biso mean: 49.3 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 2.25→30 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints NCS | NCS model details: NONE | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | Resolution: 2.25→2.39 Å / Rfactor Rfree error: 0.02 / Total num. of bins used: 6
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Xplor file |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Software | *PLUS Name:  X-PLOR / Version: 0.3 / Classification: refinement X-PLOR / Version: 0.3 / Classification: refinement | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refinement | *PLUS Highest resolution: 2.25 Å / Lowest resolution: 30 Å / σ(F): 0 / % reflection Rfree: 10.1 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Solvent computation | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Displacement parameters | *PLUS | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| Refine LS restraints | *PLUS
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| LS refinement shell | *PLUS Rfactor obs: 0.461 |
 Movie
Movie Controller
Controller











 PDBj
PDBj