+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1qky | ||||||
|---|---|---|---|---|---|---|---|
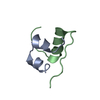
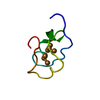
| タイトル | Solution structure of PI7, a non toxic peptide isolated from the scorpion Pandinus Imperator. | ||||||
 要素 要素 | TOXIN 7 FROM PANDINUS IMPERATOR | ||||||
 キーワード キーワード | NEUROTOXIN | ||||||
| 機能・相同性 | Scorpion short chain toxin, potassium channel inhibitor / Scorpion short toxin, BmKK2 / Knottin, scorpion toxin-like superfamily / ion channel inhibitor activity / toxin activity / extracellular region / Potassium channel toxin alpha-KTx 6.5 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  PANDINUS IMPERATOR (サソリ) PANDINUS IMPERATOR (サソリ) | ||||||
| 手法 | 溶液NMR / simulated annealing | ||||||
 データ登録者 データ登録者 | Delepierre, M. / Prochnicka-Chalufour, A. / Boisbouvier, J. / Possani, L.D. | ||||||
 引用 引用 |  ジャーナル: Biochemistry / 年: 1999 ジャーナル: Biochemistry / 年: 1999タイトル: Pi7, an orphan peptide from the scorpion Pandinus imperator: a 1H-NMR analysis using a nano-NMR Probe. 著者: Delepierre, M. / Prochnicka-Chalufour, A. / Boisbouvier, J. / Possani, L.D. #1: ジャーナル: Toxicon / 年: 1998 タイトル: Two Similar Peptides from the Venom of the Scorpion Pandinus Imperator, One Highly Effective Blocker and the Other Inactive on K+ Channels 著者: Olamendi-Portugal, T. / Gomez-Lagunas, F. / Gurrola, G.B. / Possani, L.D. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1qky.cif.gz 1qky.cif.gz | 160.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1qky.ent.gz pdb1qky.ent.gz | 132.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1qky.json.gz 1qky.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1qky_validation.pdf.gz 1qky_validation.pdf.gz | 380.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1qky_full_validation.pdf.gz 1qky_full_validation.pdf.gz | 447.8 KB | 表示 | |
| XML形式データ |  1qky_validation.xml.gz 1qky_validation.xml.gz | 10 KB | 表示 | |
| CIF形式データ |  1qky_validation.cif.gz 1qky_validation.cif.gz | 16.3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/qk/1qky https://data.pdbj.org/pub/pdb/validation_reports/qk/1qky ftp://data.pdbj.org/pub/pdb/validation_reports/qk/1qky ftp://data.pdbj.org/pub/pdb/validation_reports/qk/1qky | HTTPS FTP |
-関連構造データ
| 類似構造データ | |
|---|---|
| その他のデータベース |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 4297.019 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  PANDINUS IMPERATOR (サソリ) / Secretion: VENOM / 参照: UniProt: P58490*PLUS PANDINUS IMPERATOR (サソリ) / Secretion: VENOM / 参照: UniProt: P58490*PLUS |
|---|---|
| Has protein modification | Y |
| 配列の詳細 | THE PEPTIDE STUDIED HERE IS RELATED IN SEQUENCE TO THE POTASSIUM CHANNEL BLOCKING TOXINS, HOWEVER ...THE PEPTIDE STUDIED HERE IS RELATED IN SEQUENCE TO THE POTASSIUM CHANNEL BLOCKING TOXINS, HOWEVER THE ALIGNMENT IS ONLY 60 PERCENT FOR THE PEPTIDES: SCKA_PANIM P55927 (47 AA) SCKB_PANIM P55928 (35 AA) SCKG_PANIM Q10726 (35 AA) 1QKY DEAIRCTGTK |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験 | タイプ: NOESY |
| NMR実験の詳細 | Text: THE STRUCTURE OF THE NATURAL TOXIN EXTRACTED FROM THE SCORPION VENOM WAS DETERMINED BY HOMONUCLEAR NMR AT 500 MHZ FROM NANOMOLE AMOUNT OF COMPOUND. |
- 試料調製
試料調製
| 詳細 | 内容: H2O/D2O |
|---|---|
| 試料状態 | pH: 3.5 / 圧: 1 atm / 温度: 308 K |
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター | タイプ: Varian UNITY / 製造業者: Varian / モデル: UNITY / 磁場強度: 500 MHz |
|---|
- 解析
解析
| NMR software |
| ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: simulated annealing / ソフトェア番号: 1 詳細: THE STRUCTURE WAS CALCULATED USING THE PROGRAM DYANA. TWENTY FIVE BEST DYANA CONFORMERS WERE SUBMITTED TO RESTRAINED ENERGY MINIMIZATION IN A SHELL OF 584 WATER MOLECULES USING THE PROGRAM ...詳細: THE STRUCTURE WAS CALCULATED USING THE PROGRAM DYANA. TWENTY FIVE BEST DYANA CONFORMERS WERE SUBMITTED TO RESTRAINED ENERGY MINIMIZATION IN A SHELL OF 584 WATER MOLECULES USING THE PROGRAM OPAL. COORDINATES FOR THE WATER ARE NOT CONTAINED IN THIS ENTRY. FOR FURTHER DETAILS ON THE REFINEMENT SEE THE JRNL CITATION ABOVE. | ||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: LEAST RESTRAINT VIOLATION, STRUCTURE QUALITY ACCORDING TO PROCHECK 計算したコンフォーマーの数: 200 / 登録したコンフォーマーの数: 14 |
 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj

