+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1q9b | ||||||
|---|---|---|---|---|---|---|---|
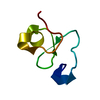
| タイトル | CRYSTAL STRUCTURE ANALYSIS OF Hev b 6.02 (HEVEIN) AT 1.5 ANGSTROMS RESOLUTION | ||||||
 要素 要素 | Hevein | ||||||
 キーワード キーワード | ALLERGEN / LECTIN / AGGLUTININ-TOXIN MOTIF | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報chitin binding / RNA nuclease activity / defense response to fungus / defense response to bacterium 類似検索 - 分子機能 | ||||||
| 生物種 |  | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.5 Å 分子置換 / 解像度: 1.5 Å | ||||||
 データ登録者 データ登録者 | Rodriguez-Romero, A. / Hernandez-Santoyo, A. | ||||||
 引用 引用 |  ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2004 ジャーナル: Biochem.Biophys.Res.Commun. / 年: 2004タイトル: Insights into a conformational epitope of Hev b 6.02 (hevein). 著者: Reyes-Lopez, C.A. / Hernandez-Santoyo, A. / Pedraza-Escalona, M. / Mendoza, G. / Hernandez-Arana, A. / Rodriguez-Romero, A. #1:  ジャーナル: FEBS Lett. / 年: 1991 ジャーナル: FEBS Lett. / 年: 1991タイトル: Crystal Structure of Hevein at 2.8 A Resolution 著者: Rodriguez-Romero, A. / Ravichandran, K.G. / Soriano-Garcia, M. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1q9b.cif.gz 1q9b.cif.gz | 32.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1q9b.ent.gz pdb1q9b.ent.gz | 21.8 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1q9b.json.gz 1q9b.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1q9b_validation.pdf.gz 1q9b_validation.pdf.gz | 436.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1q9b_full_validation.pdf.gz 1q9b_full_validation.pdf.gz | 436.9 KB | 表示 | |
| XML形式データ |  1q9b_validation.xml.gz 1q9b_validation.xml.gz | 5 KB | 表示 | |
| CIF形式データ |  1q9b_validation.cif.gz 1q9b_validation.cif.gz | 6.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/q9/1q9b https://data.pdbj.org/pub/pdb/validation_reports/q9/1q9b ftp://data.pdbj.org/pub/pdb/validation_reports/q9/1q9b ftp://data.pdbj.org/pub/pdb/validation_reports/q9/1q9b | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1hevS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||||
| 単位格子 |
|
- 要素
要素
| #1: タンパク質・ペプチド | 分子量: 4731.166 Da / 分子数: 1 / 由来タイプ: 天然 / 由来: (天然)  | ||||
|---|---|---|---|---|---|
| #2: 化合物 | | #3: 水 | ChemComp-HOH / | Has protein modification | Y | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 1.91 Å3/Da / 溶媒含有率: 35.5 % | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 291 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 7.1 詳細: MPD, Tris-HCl, pH 7.10, VAPOR DIFFUSION, SITTING DROP, temperature 291K | ||||||||||||||||||
| 結晶化 | *PLUS 温度: 18 ℃ / pH: 7.1 / 手法: 蒸気拡散法, シッティングドロップ法 | ||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 277 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  SSRL SSRL  / ビームライン: BL7-1 / 波長: 1.08 / 波長: 1.08 Å / ビームライン: BL7-1 / 波長: 1.08 / 波長: 1.08 Å |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 1999年1月15日 |
| 放射 | モノクロメーター: Si / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.08 Å / 相対比: 1 |
| 反射 | 解像度: 1.5→15 Å / Num. all: 7678 / Num. obs: 7332 / % possible obs: 95.5 % / 冗長度: 4.8 % / Biso Wilson estimate: 13.9 Å2 / Rsym value: 0.05 / Net I/σ(I): 6.7 |
| 反射 シェル | 解像度: 1.5→1.53 Å / 冗長度: 4.6 % / Mean I/σ(I) obs: 12.3 / Num. unique all: 635 / Rsym value: 0.046 / % possible all: 97.2 |
| 反射 | *PLUS 最高解像度: 1.5 Å / 最低解像度: 15 Å / Num. obs: 7113 / 冗長度: 4.7 % / Num. measured all: 57822 / Rmerge(I) obs: 0.05 |
- 解析
解析
| ソフトウェア |
| |||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB ENTRY 1HEV 解像度: 1.5→9.5 Å / Num. parameters: 3772 / Num. restraintsaints: 4884 / 交差検証法: FREE R / σ(F): 0 / 立体化学のターゲット値: ENGH AND HUBER 詳細: THE ALTERNATE CONFORMATIONS EVENTUALLY REDUCED FREE R FROM 21.19-19.20%. INTRODUCTION OF ANISOTROPIC REFINEMENT REDUCED FREE R FROM 19.20- 16.01%. DISCRETELY DISORDERED HETEROGENS: 2, MPD 101 ...詳細: THE ALTERNATE CONFORMATIONS EVENTUALLY REDUCED FREE R FROM 21.19-19.20%. INTRODUCTION OF ANISOTROPIC REFINEMENT REDUCED FREE R FROM 19.20- 16.01%. DISCRETELY DISORDERED HETEROGENS: 2, MPD 101 AND MPD 102; DISCRETELY DISORDERED RESIDUES: 6, GLU 1, LEU 11, PRO 13, SER 26, GLU 29, PRO 33. ANISOTROPIC SCALING APPLIED BY THE METHOD OF PARKIN, MOEZZI & HOPE, J.APPL.CRYST.28(1995)53-56
| |||||||||||||||||||||||||||||||||
| 溶媒の処理 | 溶媒モデル: MOEWS & KRETSINGER, J.MOL.BIOL.91(1973)201-228 | |||||||||||||||||||||||||||||||||
| Refine analyze | Num. disordered residues: 8 / Occupancy sum hydrogen: 268 / Occupancy sum non hydrogen: 379 | |||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 1.5→9.5 Å
| |||||||||||||||||||||||||||||||||
| 拘束条件 |
| |||||||||||||||||||||||||||||||||
| LS精密化 シェル | 解像度: 1.5→1.53 Å / Num. reflection Rfree: 63 / Num. reflection obs: 635 | |||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: SHELXL / バージョン: 97 / 分類: refinement | |||||||||||||||||||||||||||||||||
| 精密化 | *PLUS % reflection Rfree: 10 % / Rfactor Rwork: 0.128 | |||||||||||||||||||||||||||||||||
| 溶媒の処理 | *PLUS | |||||||||||||||||||||||||||||||||
| 原子変位パラメータ | *PLUS | |||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj


