+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1oj5 | ||||||
|---|---|---|---|---|---|---|---|
| タイトル | Crystal structure of the Nco-A1 PAS-B domain bound to the STAT6 transactivation domain LXXLL motif | ||||||
 要素 要素 |
| ||||||
 キーワード キーワード | TRANSCRIPTIONAL COACTIVATOR / COMPLEX / LXXLL MOTIF / TRANSCRIPTIONAL REGULATION / STAT6 / PAS DOMAIN / IL-4 STAT | ||||||
| 機能・相同性 |  機能・相同性情報 機能・相同性情報regulation of mast cell proliferation / NR1H2 & NR1H3 regulate gene expression to control bile acid homeostasis / NR1H3 & NR1H2 regulate gene expression linked to cholesterol transport and efflux / SUMOylation of transcription cofactors / mammary gland morphogenesis / Recycling of bile acids and salts / Synthesis of bile acids and bile salts / positive regulation of isotype switching to IgE isotypes / negative regulation of type 2 immune response / Synthesis of bile acids and bile salts via 7alpha-hydroxycholesterol ...regulation of mast cell proliferation / NR1H2 & NR1H3 regulate gene expression to control bile acid homeostasis / NR1H3 & NR1H2 regulate gene expression linked to cholesterol transport and efflux / SUMOylation of transcription cofactors / mammary gland morphogenesis / Recycling of bile acids and salts / Synthesis of bile acids and bile salts / positive regulation of isotype switching to IgE isotypes / negative regulation of type 2 immune response / Synthesis of bile acids and bile salts via 7alpha-hydroxycholesterol / Synthesis of bile acids and bile salts via 27-hydroxycholesterol / Endogenous sterols / HATs acetylate histones / T-helper 1 cell lineage commitment / STAT6-mediated induction of chemokines / Regulation of lipid metabolism by PPARalpha / Cytoprotection by HMOX1 / interleukin-4-mediated signaling pathway / Estrogen-dependent gene expression / isotype switching to IgE isotypes / labyrinthine layer morphogenesis / positive regulation of transcription from RNA polymerase II promoter by galactose / regulation of thyroid hormone receptor signaling pathway / positive regulation of female receptivity / mammary gland epithelial cell proliferation / male mating behavior / hypothalamus development / cellular response to Thyroglobulin triiodothyronine / progesterone receptor signaling pathway / growth hormone receptor signaling pathway via JAK-STAT / cell surface receptor signaling pathway via JAK-STAT / response to retinoic acid / estrous cycle / nuclear retinoid X receptor binding / histone acetyltransferase activity / histone acetyltransferase / estrogen receptor signaling pathway / lactation / positive regulation of adipose tissue development / peroxisome proliferator activated receptor signaling pathway / positive regulation of neuron differentiation / regulation of cellular response to insulin stimulus / Downstream signal transduction / response to progesterone / cerebellum development / nuclear estrogen receptor binding / RNA polymerase II transcription regulatory region sequence-specific DNA binding / hippocampus development / mRNA transcription by RNA polymerase II / defense response / response to peptide hormone / cerebral cortex development / RNA polymerase II transcription regulator complex / transcription coactivator binding / male gonad development / cytokine-mediated signaling pathway / response to estradiol / regulation of cell population proliferation / positive regulation of cold-induced thermogenesis / DNA-binding transcription activator activity, RNA polymerase II-specific / Interleukin-4 and Interleukin-13 signaling / protein phosphatase binding / transcription regulator complex / DNA-binding transcription factor activity, RNA polymerase II-specific / transcription coactivator activity / protein dimerization activity / positive regulation of apoptotic process / RNA polymerase II cis-regulatory region sequence-specific DNA binding / DNA-binding transcription factor activity / chromatin binding / regulation of transcription by RNA polymerase II / chromatin / positive regulation of DNA-templated transcription / protein-containing complex binding / negative regulation of transcription by RNA polymerase II / positive regulation of transcription by RNA polymerase II / DNA binding / nucleoplasm / identical protein binding / nucleus / plasma membrane / cytoplasm / cytosol 類似検索 - 分子機能 | ||||||
| 生物種 |   HOMO SAPIENS (ヒト) HOMO SAPIENS (ヒト) | ||||||
| 手法 |  X線回折 / OTHER / 解像度: 2.21 Å X線回折 / OTHER / 解像度: 2.21 Å | ||||||
 データ登録者 データ登録者 | Razeto, A. / Ramakrishnan, V. / Giller, K. / Lakomek, N. / Carlomagno, T. / Griesinger, C. / Lodrini, M. / Litterst, C.M. / Pftizner, E. / Becker, S. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2004 ジャーナル: J.Mol.Biol. / 年: 2004タイトル: Structure of the Ncoa-1/Src-1 Pas-B Domain Bound to the Lxxll Motif of the Stat6 Transactivation Domain 著者: Razeto, A. / Ramakrishnan, V. / Litterst, C.M. / Giller, K. / Griesinger, C. / Carlomagno, T. / Lakomek, N. / Heimburg, T. / Lodrini, M. / Pfitzner, E. / Becker, S. #1: ジャーナル: J.Biol.Chem. / 年: 2002 タイトル: An Lxxll Motif in the Transactivation Domain of Stat6 Mediates Recruitment of Ncoa-1/Src-1 著者: Litterst, C.M. / Pfitzner, E. #2: ジャーナル: J.Biol.Chem. / 年: 2001 タイトル: Transcriptional Activation by Stat6 Requires the Direct Interaction with Ncoa-1 著者: Litterst, C.M. / Pfitzner, E. | ||||||
| 履歴 |
| ||||||
| Remark 650 | HELIX DETERMINATION METHOD: AUTHOR PROVIDED. | ||||||
| Remark 700 | SHEET DETERMINATION METHOD: AUTHOR PROVIDED. |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1oj5.cif.gz 1oj5.cif.gz | 40.4 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1oj5.ent.gz pdb1oj5.ent.gz | 27.6 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1oj5.json.gz 1oj5.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1oj5_validation.pdf.gz 1oj5_validation.pdf.gz | 419.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1oj5_full_validation.pdf.gz 1oj5_full_validation.pdf.gz | 419.4 KB | 表示 | |
| XML形式データ |  1oj5_validation.xml.gz 1oj5_validation.xml.gz | 8.3 KB | 表示 | |
| CIF形式データ |  1oj5_validation.cif.gz 1oj5_validation.cif.gz | 11.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/oj/1oj5 https://data.pdbj.org/pub/pdb/validation_reports/oj/1oj5 ftp://data.pdbj.org/pub/pdb/validation_reports/oj/1oj5 ftp://data.pdbj.org/pub/pdb/validation_reports/oj/1oj5 | HTTPS FTP |
-関連構造データ
| 関連構造データ | |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 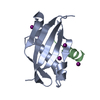
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 |
| ||||||||
| 単位格子 |
| ||||||||
| 詳細 | THIS BIOMOLECULE IS A HETERODIMERIC COMPLEX OF A PROTEINCHAIN WITH A PEPTIDE |
- 要素
要素
| #1: タンパク質 | 分子量: 14811.774 Da / 分子数: 1 / 断片: NCO-A1 PAS-B DOMAIN, RESIDUES 257-385 / 変異: YES / 由来タイプ: 組換発現 / 由来: (組換発現)   | ||||||
|---|---|---|---|---|---|---|---|
| #2: タンパク質・ペプチド | 分子量: 1610.865 Da / 分子数: 1 / 断片: STAT6 LXXLL MOTIF, RESIDUES 795-808 / 由来タイプ: 合成 / 由来: (合成)  HOMO SAPIENS (ヒト) / 参照: UniProt: P42226 HOMO SAPIENS (ヒト) / 参照: UniProt: P42226 | ||||||
| #3: 化合物 | ChemComp-IOD / #4: 水 | ChemComp-HOH / | 構成要素の詳細 | ENGINEERED | 配列の詳細 | RESIDUES A254-A256 ARE FROM THE VECTOR PET16BTEV | |
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 2.5 Å3/Da / 溶媒含有率: 50 % | |||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | pH: 7 / 詳細: 20 % PEG3350, 0.2 M LICL, pH 7.00 | |||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 20 ℃ / pH: 7 / 手法: 蒸気拡散法, ハンギングドロップ法 | |||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  回転陽極 / タイプ: SIEMENS M18X / 波長: 1.5418 回転陽極 / タイプ: SIEMENS M18X / 波長: 1.5418 |
| 検出器 | タイプ: MARRESEARCH / 検出器: IMAGE PLATE / 日付: 2003年3月24日 / 詳細: OSMIC MIRRORS CMF12-38CU6 |
| 放射 | モノクロメーター: OSMIC MIRRORS CMF12-38CU6 / プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.5418 Å / 相対比: 1 |
| 反射 | 解像度: 2.21→19.21 Å / Num. obs: 8138 / % possible obs: 99.7 % / 冗長度: 16.8 % / Biso Wilson estimate: 28.7 Å2 / Rmerge(I) obs: 0.089 / Net I/σ(I): 29.9 |
| 反射 シェル | 解像度: 2.21→2.25 Å / 冗長度: 16 % / Rmerge(I) obs: 0.3254 / Mean I/σ(I) obs: 8.71 / % possible all: 98.1 |
| 反射 | *PLUS 最高解像度: 2.21 Å / 最低解像度: 19.2 Å / 冗長度: 16.8 % / Num. measured all: 137915 / Rmerge(I) obs: 0.089 |
| 反射 シェル | *PLUS % possible obs: 98.1 % / 冗長度: 16 % / Rmerge(I) obs: 0.325 / Mean I/σ(I) obs: 8.7 |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法: OTHER / 解像度: 2.21→19.21 Å / Cor.coef. Fo:Fc: 0.953 / Cor.coef. Fo:Fc free: 0.928 / SU B: 2.825 / SU ML: 0.075 / 交差検証法: THROUGHOUT / ESU R: 0.195 / ESU R Free: 0.178 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. THE LAST RUN OF REFINEMENT WAS PERFORMED WITH ALL THE REFLECTIONS. THE R FREE REPORTED ABOVE IS REFERRED TO THE PREVIOUS RUN OF REFINEMENT. ...詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS. THE LAST RUN OF REFINEMENT WAS PERFORMED WITH ALL THE REFLECTIONS. THE R FREE REPORTED ABOVE IS REFERRED TO THE PREVIOUS RUN OF REFINEMENT. SIDE CHAINS WITH POOR ELECTRON DENSITY WERE ASSIGNED OCCUPANCY 0.5
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶媒の処理 | イオンプローブ半径: 0.8 Å / 減衰半径: 0.8 Å / VDWプローブ半径: 1.4 Å / 溶媒モデル: BABINET MODEL PLUS MASK | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 32.92 Å2
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.21→19.21 Å
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
|
 ムービー
ムービー コントローラー
コントローラー















 PDBj
PDBj




















