+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1o15 | ||||||
|---|---|---|---|---|---|---|---|
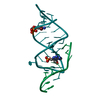
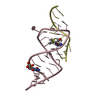
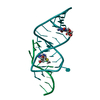
| タイトル | THEOPHYLLINE-BINDING RNA IN COMPLEX WITH THEOPHYLLINE, NMR, REGULARIZED MEAN STRUCTURE, REFINEMENT WITH TORSION ANGLE AND BASE-BASE POSITIONAL DATABASE POTENTIALS AND DIPOLAR COUPLINGS | ||||||
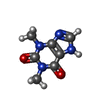
 要素 要素 | THEOPHYLLINE-BINDING RNA | ||||||
 キーワード キーワード | RNA / RIBONUCLEIC ACID | ||||||
| 機能・相同性 | THEOPHYLLINE / RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 | 溶液NMR | ||||||
 データ登録者 データ登録者 | Clore, G.M. / Kuszewski, J. | ||||||
 引用 引用 |  ジャーナル: J.Am.Chem.Soc. / 年: 2003 ジャーナル: J.Am.Chem.Soc. / 年: 2003タイトル: Improving the Accuracy of NMR Structures of RNA by Means of Conformational Database Potentials of Mean Force as Assessed by Complete Dipolar Coupling Cross-Validation 著者: Clore, G.M. / Kuszewski, J. #1:  ジャーナル: Nat.Struct.Biol. / 年: 1997 ジャーナル: Nat.Struct.Biol. / 年: 1997タイトル: Interlocking Structural Motifs Mediate Molecular Discrimination by a Theophylline-Binding RNA 著者: Zimmermann, G.R. / Jenison, R.D. / Wick, C.L. / Simorre, J.P. / Pardi, A. #2:  ジャーナル: J.Am.Chem.Soc. / 年: 2001 ジャーナル: J.Am.Chem.Soc. / 年: 2001タイトル: Refinement of Local and Long Range Structural Order in Theophylline-Binding RNA Using Using 13C-1H Residual Dipolar Couplings and Restrained Molecular Dynamics. 著者: Sibille, N. / Pardi, A. / Simorre, J.P. / Blackledge, M. #3:  ジャーナル: J.Am.Chem.Soc. / 年: 2001 ジャーナル: J.Am.Chem.Soc. / 年: 2001タイトル: Improving the Accuracy of NMR Structures of DNA by Means of a Database Potential of Mean Force Describing Base-Base Positional Interactions. 著者: Kuszewski, J. / Schwieters, C. / Clore, G.M. | ||||||
| 履歴 |
| ||||||
| Remark 8 | THE RESTRAINTS USED FOR DETERMINATION OF THIS STRUCTURE ARE SUMMARIZED AS FOLLOWS: DISTANCE ... THE RESTRAINTS USED FOR DETERMINATION OF THIS STRUCTURE ARE SUMMARIZED AS FOLLOWS: DISTANCE RESTRAINTS 30 INTRARESIDUE NOES 86 SEQUENTIAL NOES 17 MEDIUM RANGE (1 < |i-j|<5) NOES 90 LONG RANGE (|i-j|>=5) NOES 52 DISTANCES FOR 8 W-C BASE PAIRS and 1 G-U WOBBLE PAIR TOTAL: 223 LOOSE TORSION ANGLE RESTRAINTS 31 DELTA 33 CHI 9 ALPHA, 9 BETA, 10 GAMMA, 9 EPSILON AND 9 ZETA TOTAL: 110. 13C-1H DIPOLAR COUPLINGS 55 SUGAR 46 BASE TOTAL 101 |
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1o15.cif.gz 1o15.cif.gz | 33 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1o15.ent.gz pdb1o15.ent.gz | 23.3 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1o15.json.gz 1o15.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1o15_validation.pdf.gz 1o15_validation.pdf.gz | 336.3 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1o15_full_validation.pdf.gz 1o15_full_validation.pdf.gz | 336.4 KB | 表示 | |
| XML形式データ |  1o15_validation.xml.gz 1o15_validation.xml.gz | 2.6 KB | 表示 | |
| CIF形式データ |  1o15_validation.cif.gz 1o15_validation.cif.gz | 3 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/o1/1o15 https://data.pdbj.org/pub/pdb/validation_reports/o1/1o15 ftp://data.pdbj.org/pub/pdb/validation_reports/o1/1o15 ftp://data.pdbj.org/pub/pdb/validation_reports/o1/1o15 | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 10638.409 Da / 分子数: 1 / 由来タイプ: 合成 詳細: CONSENSUS SEQUENCE OF THE THEOPHYLLINE BINDING RNA APTAMER FLANKED BY A FOUR BASE-PAIR STEM, A THREE BASE-PAIR STEM, AND CAPPED BY A GAAA TETRALOOP |
|---|---|
| #2: 化合物 | ChemComp-TEP / |
-実験情報
-実験
| 実験 | 手法: 溶液NMR |
|---|---|
| NMR実験の詳細 | Text: THE DIPOLAR COUPLINGS WERE DIVIDED INTO 10 PAIRS OF WORKING AND TEST DATA SETS CHOSEN AT RANDOM AND PARTITIONED IN A RATIO OF 70% (WORKING) AND 30% (TEST). 25 SIMULATED ANNEALING STRUCTURES ...Text: THE DIPOLAR COUPLINGS WERE DIVIDED INTO 10 PAIRS OF WORKING AND TEST DATA SETS CHOSEN AT RANDOM AND PARTITIONED IN A RATIO OF 70% (WORKING) AND 30% (TEST). 25 SIMULATED ANNEALING STRUCTURES WERE CALCULATED FOR EACH PAIR, RESULTING IN A TOTAL OF 250 STRUCTURES. THE STRUCTURE REPORTED HERE IS OBTAINED BY RESTRAINED REGULARIZED OF THE MEAN COORDINATES OF THE ENSEMBLE OF 250 INDIVIDUAL SIMULATED ANNEALING STRUCTURES CALCULATED WITH COMPLETE DIPOLAR COUPLING CROSS-VALIDATION. FOR THE ENSEMBLE OF 250 STRUCTURES THE WORKING DIPOLAR COUPLING R-FACTOR IS 8.6+/-0.5% AND THE FREE DIPOLAR COUPLING R-FACTOR IS 26.8+/-2.8%. |
- 試料調製
試料調製
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
|---|
-NMR測定
| 放射 | プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M |
|---|---|
| 放射波長 | 相対比: 1 |
- 解析
解析
| NMR software | 名称:  X-PLOR NIH / バージョン: (HTTP://NMR.CIT.NIH.GOV/XPLOR_NIH) / 開発者: CLORE, KUSZEWSKI, SCHWIETERS, TJANDRA / 分類: 精密化 X-PLOR NIH / バージョン: (HTTP://NMR.CIT.NIH.GOV/XPLOR_NIH) / 開発者: CLORE, KUSZEWSKI, SCHWIETERS, TJANDRA / 分類: 精密化 |
|---|---|
| 精密化 | ソフトェア番号: 1 詳細: THE STRUCTURE IS BASED ON A TOTAL OF 223 NOE, 52 DISTANCES FOR WATSON-CRICK HYDROGEN BONDS, AND 110 TORSION ANGLE RESTRAINTS, 101 CH DIPOLAR COUPLING RESTRAINTS. THE EXPERIMENTAL RESTRAINTS ...詳細: THE STRUCTURE IS BASED ON A TOTAL OF 223 NOE, 52 DISTANCES FOR WATSON-CRICK HYDROGEN BONDS, AND 110 TORSION ANGLE RESTRAINTS, 101 CH DIPOLAR COUPLING RESTRAINTS. THE EXPERIMENTAL RESTRAINTS ARE ESSENTIALLY THE SAME AS THOSE LISTED IN N.SIBILLE,A.PARDI,J.P.SIMORRE,M.BLACKLEDGE J.AM.CHEM.SOC. V 123, 12135 2001. THE NON-BONDED CONTACTS ARE REPRESENTED BY A QUARTIC VAN DER WAALS REPULSION TERM, A BASE-BASE POSITIONING DATABASE POTENTIAL OF MEAN FORCE, AND A TORSION ANGLE DATABASE POTENTIAL OF MEAN FORCE. |
| NMRアンサンブル | コンフォーマー選択の基準: RESTRAINED REGULARIZED MEAN STRUCTURE 計算したコンフォーマーの数: 250 / 登録したコンフォーマーの数: 1 |
 ムービー
ムービー コントローラー
コントローラー










 PDBj
PDBj