+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1mnx | ||||||
|---|---|---|---|---|---|---|---|
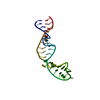
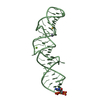
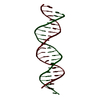
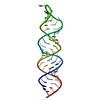
| タイトル | The Solution Structure of the Loop E Region of the 5S rRNA from Spinach Chloroplasts. | ||||||
 要素 要素 | Loop E from 5S rRNA | ||||||
 キーワード キーワード | RNA / Loop E / 5S rRNA | ||||||
| 機能・相同性 | RNA / RNA (> 10) 機能・相同性情報 機能・相同性情報 | ||||||
| 生物種 |  Spinacia oleracea (ホウレンソウ) Spinacia oleracea (ホウレンソウ) | ||||||
| 手法 | 溶液NMR / torsion angle dynamics, molecular dynamics | ||||||
| Model type details | minimized average | ||||||
 データ登録者 データ登録者 | Vallurupalli, P. / Moore, P.B. | ||||||
 引用 引用 |  ジャーナル: J.Mol.Biol. / 年: 2003 ジャーナル: J.Mol.Biol. / 年: 2003タイトル: The Solution Structure of the Loop E Region of the 5S rRNA from Spinach Chloroplasts 著者: Vallurupalli, P. / Moore, P.B. #1:  ジャーナル: RNA / 年: 1998 ジャーナル: RNA / 年: 1998タイトル: The 5S rRNA loop E: Chemical probing and phylogenetic data versus crystal structure 著者: Leontis, N.B. / Westhof, E. #2:  ジャーナル: Structure / 年: 1997 ジャーナル: Structure / 年: 1997タイトル: The loop E-loop D region of Escherichia coli 5S rRNA: the solution structure reveals an unusual loop that may be important for binding ribosomal proteins. 著者: Dallas, A. / Moore, P.B. #3:  ジャーナル: Cell(Cambridge,Mass.) / 年: 1997 ジャーナル: Cell(Cambridge,Mass.) / 年: 1997タイトル: Metals, Motifs, and Recognition in the Crystal Structure of a 5S rRNA domain 著者: Correll, C. / Freeborn, B. / Moore, P.B. / Steitz, T.A. #4:  ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2000 ジャーナル: Proc.Natl.Acad.Sci.USA / 年: 2000タイトル: Structure of Escherichia coli ribosomal protein L25 complexed with a 5S rRNA fragment at 1.8-A resolution 著者: Lu, M. / Steitz, T.A. #5:  ジャーナル: Embo J. / 年: 1999 ジャーナル: Embo J. / 年: 1999タイトル: The NMR structure of the 5S rRNA E-domain-protein L25 complex shows preformed and induced recognition 著者: Stoldt, M. / Wohnert, J. / Ohlenschlager, O. / Gorlach, M. / Brown, L.R. #6:  ジャーナル: Biochemistry / 年: 1988 ジャーナル: Biochemistry / 年: 1988タイトル: Higher order structure of chloroplastic 5S ribosomal RNA from spinach. 著者: Romby, P. / Westhof, E. / Toukifimpa, R. / Mache, R. / Ebel, J.P. / Ehresmann, C. / Ehresmann, B. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1mnx.cif.gz 1mnx.cif.gz | 350.7 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1mnx.ent.gz pdb1mnx.ent.gz | 296.7 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1mnx.json.gz 1mnx.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1mnx_validation.pdf.gz 1mnx_validation.pdf.gz | 323.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1mnx_full_validation.pdf.gz 1mnx_full_validation.pdf.gz | 395.3 KB | 表示 | |
| XML形式データ |  1mnx_validation.xml.gz 1mnx_validation.xml.gz | 6.3 KB | 表示 | |
| CIF形式データ |  1mnx_validation.cif.gz 1mnx_validation.cif.gz | 12.6 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/mn/1mnx https://data.pdbj.org/pub/pdb/validation_reports/mn/1mnx ftp://data.pdbj.org/pub/pdb/validation_reports/mn/1mnx ftp://data.pdbj.org/pub/pdb/validation_reports/mn/1mnx | HTTPS FTP |
-関連構造データ
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| |||||||||
|---|---|---|---|---|---|---|---|---|---|---|
| 1 |
| |||||||||
| NMR アンサンブル |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 13667.229 Da / 分子数: 1 / 由来タイプ: 合成 詳細: RNA was in vitro transcribed using T7 RNA polymerase from a linearized plasmid containing the required sequence. The sequence is naturally found in Spinacia oleracea (Spinach) cholorplasts. 由来: (合成)  Spinacia oleracea (ホウレンソウ) Spinacia oleracea (ホウレンソウ) |
|---|
-実験情報
-実験
| 実験 | 手法: 溶液NMR | ||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| NMR実験 |
| ||||||||||||
| NMR実験の詳細 | Text: This structure was determined using data obtained from homonuclear and heteronuclear experiments. |
- 試料調製
試料調製
| 詳細 | 内容: RNA molecule (1mM-2mM) in 50 mM NaCl, 2.5mM Cacodylate, 0.1mM EDTA, pH 6.0 溶媒系: H2O, D2O | |||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 試料状態 |
| |||||||||||||||
| 結晶化 | *PLUS 手法: other / 詳細: NMR |
-NMR測定
| NMRスペクトロメーター |
|
|---|
- 解析
解析
| NMR software |
| ||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 手法: torsion angle dynamics, molecular dynamics / ソフトェア番号: 1 詳細: Distance, Dihedral, Planarity restraints were used in the first round, followed by dipolar couplings in the second round (See paper for details) | ||||||||||||||||
| 代表構造 | 選択基準: minimized average structure | ||||||||||||||||
| NMRアンサンブル | コンフォーマー選択の基準: structures with the least restraint violations,structures with the lowest energy 計算したコンフォーマーの数: 65 / 登録したコンフォーマーの数: 13 |
 ムービー
ムービー コントローラー
コントローラー









 PDBj
PDBj





























 HSQC
HSQC