| 登録情報 | データベース: PDB / ID: 1kmt
|
|---|
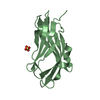
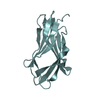
| タイトル | Crystal structure of RhoGDI Glu(154,155)Ala mutant |
|---|
 要素 要素 | Rho GDP-dissociation inhibitor 1 |
|---|
 キーワード キーワード | PROTEIN BINDING / IMMUNOGLOBULIN FOLD / BETA SANDWICH MOTIF / ISOPRENYL-BINDING |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Rho GDP-dissociation inhibitor activity / Axonal growth inhibition (RHOA activation) / Axonal growth stimulation / regulation of Rho protein signal transduction / regulation of synaptic vesicle cycle / RHOC GTPase cycle / semaphorin-plexin signaling pathway / CDC42 GTPase cycle / RHOH GTPase cycle / immunological synapse ...Rho GDP-dissociation inhibitor activity / Axonal growth inhibition (RHOA activation) / Axonal growth stimulation / regulation of Rho protein signal transduction / regulation of synaptic vesicle cycle / RHOC GTPase cycle / semaphorin-plexin signaling pathway / CDC42 GTPase cycle / RHOH GTPase cycle / immunological synapse / RHOG GTPase cycle / RHOA GTPase cycle / RAC2 GTPase cycle / Rho protein signal transduction / RAC1 GTPase cycle / GTPase activator activity / Schaffer collateral - CA1 synapse / regulation of protein localization / cytoskeleton / negative regulation of apoptotic process / extracellular exosome / nucleus / membrane / cytosol類似検索 - 分子機能 Coagulation Factor XIII, subunit A, domain 1 / Rho protein GDP-dissociation inhibitor / Rho GDP-dissociation inhibitor domain superfamily / RHO protein GDP dissociation inhibitor / Coagulation Factor XIII; Chain A, domain 1 / Distorted Sandwich / Immunoglobulin E-set / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |  Homo sapiens (ヒト) Homo sapiens (ヒト) |
|---|
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 1.3 Å 分子置換 / 解像度: 1.3 Å |
|---|
 データ登録者 データ登録者 | Mateja, A. / Devedjiev, Y. / Krowarsh, D. / Longenecker, K. / Dauter, Z. / Otlewski, J. / Derewenda, Z.S. |
|---|
 引用 引用 |  ジャーナル: Acta Crystallogr.,Sect.D / 年: 2002 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2002
タイトル: The impact of Glu-->Ala and Glu-->Asp mutations on the crystallization properties of RhoGDI: the structure of RhoGDI at 1.3 A resolution.
著者: Mateja, A. / Devedjiev, Y. / Krowarsch, D. / Longenecker, K. / Dauter, Z. / Otlewski, J. / Derewenda, Z.S. |
|---|
| 履歴 | | 登録 | 2001年12月17日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2002年12月11日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2021年10月27日 | Group: Database references / カテゴリ: database_2 / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession / _struct_ref_seq_dif.details |
|---|
| 改定 1.4 | 2024年2月14日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報 Homo sapiens (ヒト)
Homo sapiens (ヒト) X線回折 /
X線回折 /  シンクロトロン /
シンクロトロン /  分子置換 / 解像度: 1.3 Å
分子置換 / 解像度: 1.3 Å  データ登録者
データ登録者 引用
引用 ジャーナル: Acta Crystallogr.,Sect.D / 年: 2002
ジャーナル: Acta Crystallogr.,Sect.D / 年: 2002 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1kmt.cif.gz
1kmt.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1kmt.ent.gz
pdb1kmt.ent.gz PDB形式
PDB形式 1kmt.json.gz
1kmt.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード https://data.pdbj.org/pub/pdb/validation_reports/km/1kmt
https://data.pdbj.org/pub/pdb/validation_reports/km/1kmt ftp://data.pdbj.org/pub/pdb/validation_reports/km/1kmt
ftp://data.pdbj.org/pub/pdb/validation_reports/km/1kmt リンク
リンク 集合体
集合体


 要素
要素 Homo sapiens (ヒト) / 発現宿主:
Homo sapiens (ヒト) / 発現宿主: 
 X線回折 / 使用した結晶の数: 1
X線回折 / 使用した結晶の数: 1  試料調製
試料調製 シンクロトロン / サイト:
シンクロトロン / サイト:  NSLS
NSLS  / ビームライン: X9B / 波長: 0.98 Å
/ ビームライン: X9B / 波長: 0.98 Å 解析
解析 分子置換 / 解像度: 1.3→25 Å / Cor.coef. Fo:Fc: 0.975 / Cor.coef. Fo:Fc free: 0.957 / SU B: 4.457 / SU ML: 0.1 / 交差検証法: THROUGHOUT / σ(F): 1 / ESU R: 0.056 / ESU R Free: 0.055 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS
分子置換 / 解像度: 1.3→25 Å / Cor.coef. Fo:Fc: 0.975 / Cor.coef. Fo:Fc free: 0.957 / SU B: 4.457 / SU ML: 0.1 / 交差検証法: THROUGHOUT / σ(F): 1 / ESU R: 0.056 / ESU R Free: 0.055 / 立体化学のターゲット値: MAXIMUM LIKELIHOOD / 詳細: HYDROGENS HAVE BEEN ADDED IN THE RIDING POSITIONS ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj





