| 登録情報 | データベース: PDB / ID: 1k76
|
|---|
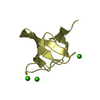
| タイトル | Solution Structure of the C-terminal Sem-5 SH3 Domain (Minimized Average Structure) |
|---|
 要素 要素 | SEX MUSCLE ABNORMAL PROTEIN 5 |
|---|
 キーワード キーワード | SIGNALING PROTEIN / all beta protein |
|---|
| 機能・相同性 |  機能・相同性情報 機能・相同性情報
Signaling by SCF-KIT / Regulation of KIT signaling / GRB2 events in ERBB2 signaling / GRB2:SOS provides linkage to MAPK signaling for Integrins / NCAM signaling for neurite out-growth / SHC-mediated cascade:FGFR1 / PI-3K cascade:FGFR1 / FRS-mediated FGFR1 signaling / SHC-mediated cascade:FGFR2 / FRS-mediated FGFR2 signaling ...Signaling by SCF-KIT / Regulation of KIT signaling / GRB2 events in ERBB2 signaling / GRB2:SOS provides linkage to MAPK signaling for Integrins / NCAM signaling for neurite out-growth / SHC-mediated cascade:FGFR1 / PI-3K cascade:FGFR1 / FRS-mediated FGFR1 signaling / SHC-mediated cascade:FGFR2 / FRS-mediated FGFR2 signaling / SHC-mediated cascade:FGFR3 / FRS-mediated FGFR3 signaling / FRS-mediated FGFR4 signaling / SHC-mediated cascade:FGFR4 / PI-3K cascade:FGFR4 / Insulin receptor signalling cascade / MET activates RAS signaling / MET activates PI3K/AKT signaling / RHOU GTPase cycle / FLT3 Signaling / PIP3 activates AKT signaling / GRB2 events in EGFR signaling / SHC1 events in EGFR signaling / PI3K events in ERBB2 signaling / Regulation of actin dynamics for phagocytic cup formation / EGFR Transactivation by Gastrin / RHO GTPases Activate WASPs and WAVEs / RAF/MAP kinase cascade / PI5P, PP2A and IER3 Regulate PI3K/AKT Signaling / Downstream signal transduction / MET activates RAP1 and RAC1 / MET receptor recycling / regulation of nematode larval development / Negative regulation of MET activity / EGFR downregulation / Cargo recognition for clathrin-mediated endocytosis / Regulation of signaling by CBL / regulation of vulval development / Clathrin-mediated endocytosis / regulation of cell projection organization / male genitalia development / COP9 signalosome / epidermal growth factor receptor binding / muscle organ development / regulation of MAPK cascade / phosphotyrosine residue binding / regulation of cell migration / epidermal growth factor receptor signaling pathway / signal transduction / nucleoplasm / plasma membrane / cytoplasm類似検索 - 分子機能 Grb2-like / SH3 Domains / SH3 domain / SH2 domain / Src homology 2 (SH2) domain profile. / Src homology 2 domains / SH2 domain / Src homology 3 domains / SH3 type barrels. / SH2 domain superfamily ...Grb2-like / SH3 Domains / SH3 domain / SH2 domain / Src homology 2 (SH2) domain profile. / Src homology 2 domains / SH2 domain / Src homology 3 domains / SH3 type barrels. / SH2 domain superfamily / SH3-like domain superfamily / Src homology 3 (SH3) domain profile. / SH3 domain / Roll / Mainly Beta類似検索 - ドメイン・相同性 |
|---|
| 生物種 |   Caenorhabditis elegans (センチュウ) Caenorhabditis elegans (センチュウ) |
|---|
| 手法 | 溶液NMR / distance geometry simulated annealing, molecular dynamics |
|---|
| Model type details | minimized average |
|---|
 データ登録者 データ登録者 | Ferreon, J. / Volk, D. / Luxon, B. / Gorenstein, D. / Hilser, V. |
|---|
 引用 引用 |  ジャーナル: Biochemistry / 年: 2003 ジャーナル: Biochemistry / 年: 2003
タイトル: Solution Structure, Dynamics and Thermodynamics of the Native State Ensemble of Sem-5 C-Terminal SH3 Domain
著者: Ferreon, J. / Volk, D. / Luxon, B. / Gorenstein, D. / Hilser, V. |
|---|
| 履歴 | | 登録 | 2001年10月18日 | 登録サイト: RCSB / 処理サイト: RCSB |
|---|
| 改定 1.0 | 2003年5月20日 | Provider: repository / タイプ: Initial release |
|---|
| 改定 1.1 | 2008年4月27日 | Group: Version format compliance |
|---|
| 改定 1.2 | 2011年7月13日 | Group: Version format compliance |
|---|
| 改定 1.3 | 2021年10月27日 | Group: Data collection / Database references / Derived calculations
カテゴリ: database_2 / pdbx_nmr_software ...database_2 / pdbx_nmr_software / pdbx_struct_assembly / pdbx_struct_oper_list / struct_ref_seq_dif
Item: _database_2.pdbx_DOI / _database_2.pdbx_database_accession ..._database_2.pdbx_DOI / _database_2.pdbx_database_accession / _pdbx_nmr_software.name / _struct_ref_seq_dif.details |
|---|
| 改定 1.4 | 2024年5月22日 | Group: Data collection / カテゴリ: chem_comp_atom / chem_comp_bond |
|---|
|
|---|
 データを開く
データを開く 基本情報
基本情報 要素
要素 キーワード
キーワード 機能・相同性情報
機能・相同性情報
 データ登録者
データ登録者 引用
引用 ジャーナル: Biochemistry / 年: 2003
ジャーナル: Biochemistry / 年: 2003 構造の表示
構造の表示 Molmil
Molmil Jmol/JSmol
Jmol/JSmol ダウンロードとリンク
ダウンロードとリンク ダウンロード
ダウンロード 1k76.cif.gz
1k76.cif.gz PDBx/mmCIF形式
PDBx/mmCIF形式 pdb1k76.ent.gz
pdb1k76.ent.gz PDB形式
PDB形式 1k76.json.gz
1k76.json.gz PDBx/mmJSON形式
PDBx/mmJSON形式 その他のダウンロード
その他のダウンロード 1k76_validation.pdf.gz
1k76_validation.pdf.gz wwPDB検証レポート
wwPDB検証レポート 1k76_full_validation.pdf.gz
1k76_full_validation.pdf.gz 1k76_validation.xml.gz
1k76_validation.xml.gz 1k76_validation.cif.gz
1k76_validation.cif.gz https://data.pdbj.org/pub/pdb/validation_reports/k7/1k76
https://data.pdbj.org/pub/pdb/validation_reports/k7/1k76 ftp://data.pdbj.org/pub/pdb/validation_reports/k7/1k76
ftp://data.pdbj.org/pub/pdb/validation_reports/k7/1k76 リンク
リンク 集合体
集合体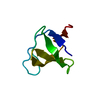
 要素
要素

 試料調製
試料調製 解析
解析 ムービー
ムービー コントローラー
コントローラー











 PDBj
PDBj HSQC
HSQC