[English] 日本語
 Yorodumi
Yorodumi- PDB-1i2y: 1.66 A STRUCTURE OF A-DUPLEX WITH BULGED ADENOSINE, SPERMINE FORM -
+ Open data
Open data
- Basic information
Basic information
| Entry | Database: PDB / ID: 1i2y | ||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Title | 1.66 A STRUCTURE OF A-DUPLEX WITH BULGED ADENOSINE, SPERMINE FORM | ||||||||||||||||||
 Components Components | DNA/RNA (5'-R(* Keywords KeywordsDNA-RNA HYBRID / adenine bulge / A-type NA / DNA/RNA hybrid | Function / homology | SPERMINE / DNA/RNA hybrid / DNA/RNA hybrid (> 10) |  Function and homology information Function and homology informationMethod |  X-RAY DIFFRACTION / X-RAY DIFFRACTION /  SYNCHROTRON / SYNCHROTRON /  MOLECULAR REPLACEMENT / Resolution: 1.66 Å MOLECULAR REPLACEMENT / Resolution: 1.66 Å  Authors AuthorsTereshko, V. / Wallace, S. / Usman, N. / Wincott, F. / Egli, M. |  Citation Citation Journal: RNA / Year: 2001 Journal: RNA / Year: 2001Title: X-ray crystallographic observation of "in-line" and "adjacent" conformations in a bulged self-cleaving RNA/DNA hybrid. Authors: Tereshko, V. / Wallace, S.T. / Usman, N. / Wincott, F.E. / Egli, M. History |
|
- Structure visualization
Structure visualization
| Structure viewer | Molecule:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- Downloads & links
Downloads & links
- Download
Download
| PDBx/mmCIF format |  1i2y.cif.gz 1i2y.cif.gz | 54.3 KB | Display |  PDBx/mmCIF format PDBx/mmCIF format |
|---|---|---|---|---|
| PDB format |  pdb1i2y.ent.gz pdb1i2y.ent.gz | 38.1 KB | Display |  PDB format PDB format |
| PDBx/mmJSON format |  1i2y.json.gz 1i2y.json.gz | Tree view |  PDBx/mmJSON format PDBx/mmJSON format | |
| Others |  Other downloads Other downloads |
-Validation report
| Arichive directory |  https://data.pdbj.org/pub/pdb/validation_reports/i2/1i2y https://data.pdbj.org/pub/pdb/validation_reports/i2/1i2y ftp://data.pdbj.org/pub/pdb/validation_reports/i2/1i2y ftp://data.pdbj.org/pub/pdb/validation_reports/i2/1i2y | HTTPS FTP |
|---|
-Related structure data
- Links
Links
- Assembly
Assembly
| Deposited unit | 
| ||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|
| 1 | 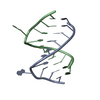
| ||||||||||
| 2 | 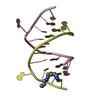
| ||||||||||
| 3 | 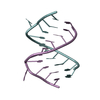
| ||||||||||
| Unit cell |
|
- Components
Components
| #1: DNA/RNA hybrid | Mass: 3471.194 Da / Num. of mol.: 6 / Source method: obtained synthetically #2: Chemical | ChemComp-SPM / | #3: Water | ChemComp-HOH / | |
|---|
-Experimental details
-Experiment
| Experiment | Method:  X-RAY DIFFRACTION / Number of used crystals: 1 X-RAY DIFFRACTION / Number of used crystals: 1 |
|---|
- Sample preparation
Sample preparation
| Crystal | Density Matthews: 2.66 Å3/Da / Density % sol: 53.77 % | |||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Crystal grow | Temperature: 298 K / Method: vapor diffusion, sitting drop / pH: 6 Details: spermine, MPD. pH 6.0, VAPOR DIFFUSION, SITTING DROP at 298 | |||||||||||||||||||||||||
| Components of the solutions |
| |||||||||||||||||||||||||
| Crystal grow | *PLUS Method: vapor diffusion, hanging drop / PH range low: 9 / PH range high: 5.5 | |||||||||||||||||||||||||
| Components of the solutions | *PLUS
|
-Data collection
| Diffraction | Mean temperature: 100 K |
|---|---|
| Diffraction source | Source:  SYNCHROTRON / Site: SYNCHROTRON / Site:  APS APS  / Beamline: 5ID-B / Wavelength: 1 Å / Beamline: 5ID-B / Wavelength: 1 Å |
| Detector | Type: MARRESEARCH / Detector: CCD / Date: Jan 11, 1998 |
| Radiation | Protocol: SINGLE WAVELENGTH / Monochromatic (M) / Laue (L): M / Scattering type: x-ray |
| Radiation wavelength | Wavelength: 1 Å / Relative weight: 1 |
| Reflection | Resolution: 1.66→20 Å / Num. all: 26418 / Num. obs: 26418 / % possible obs: 100 % / Observed criterion σ(F): 0 / Observed criterion σ(I): 0 |
| Reflection | *PLUS Highest resolution: 1.65 Å / Num. obs: 26875 / % possible obs: 97.4 % / Rmerge(I) obs: 0.042 |
- Processing
Processing
| Software |
| ||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| Refinement | Method to determine structure:  MOLECULAR REPLACEMENT / Resolution: 1.66→10 Å / Cross valid method: througout / σ(F): 0 / σ(I): 0 MOLECULAR REPLACEMENT / Resolution: 1.66→10 Å / Cross valid method: througout / σ(F): 0 / σ(I): 0
| ||||||||||||||||||||||||||||||
| Refinement step | Cycle: LAST / Resolution: 1.66→10 Å
| ||||||||||||||||||||||||||||||
| Software | *PLUS Name: CNS / Version: 1 / Classification: refinement | ||||||||||||||||||||||||||||||
| Refinement | *PLUS Lowest resolution: 10 Å / Num. reflection obs: 24925 / σ(F): 1 / % reflection Rfree: 10 % / Rfactor obs: 0.217 / Rfactor Rfree: 0.245 | ||||||||||||||||||||||||||||||
| Solvent computation | *PLUS | ||||||||||||||||||||||||||||||
| Displacement parameters | *PLUS | ||||||||||||||||||||||||||||||
| Refine LS restraints | *PLUS
| ||||||||||||||||||||||||||||||
| LS refinement shell | *PLUS Highest resolution: 1.65 Å / Lowest resolution: 1.71 Å / Rfactor Rfree: 0.281 / Rfactor Rwork: 0.285 / Num. reflection Rwork: 1169 |
 Movie
Movie Controller
Controller






 PDBj
PDBj