+ データを開く
データを開く
- 基本情報
基本情報
| 登録情報 | データベース: PDB / ID: 1hr2 | ||||||
|---|---|---|---|---|---|---|---|
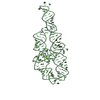
| タイトル | CRYSTAL STRUCTURE ANALYSIS OF A MUTANT P4-P6 DOMAIN (DELC209) OF TETRAHYMENA THEMOPHILA GROUP I INTRON. | ||||||
 要素 要素 | P4-P6 DELC209 MUTANT RNA RIBOZYME DOMAIN | ||||||
 キーワード キーワード | RNA / P4-P6 / C209 / RIBOZYME / TETRAHYMENA / GROUP I INTRON / RIBONUCLEIC ACID | ||||||
| 機能・相同性 | RNA / RNA (> 10) / RNA (> 100) 機能・相同性情報 機能・相同性情報 | ||||||
| 手法 |  X線回折 / X線回折 /  シンクロトロン / シンクロトロン /  分子置換 / 解像度: 2.25 Å 分子置換 / 解像度: 2.25 Å | ||||||
 データ登録者 データ登録者 | Juneau, K. / Podell, E.R. / Harrington, D.J. / Cech, T.R. | ||||||
 引用 引用 |  ジャーナル: Structure / 年: 2001 ジャーナル: Structure / 年: 2001タイトル: Structural basis of the enhanced stability of a mutant ribozyme domain and a detailed view of RNA--solvent interactions. 著者: Juneau, K. / Podell, E. / Harrington, D.J. / Cech, T.R. #1:  ジャーナル: RNA / 年: 1999 ジャーナル: RNA / 年: 1999タイトル: In-Vitro Selection of RNAs With Increased Tertiary Structure Stability 著者: Juneau, K. / Cech, T.R. #2:  ジャーナル: Science / 年: 1996 ジャーナル: Science / 年: 1996タイトル: Crystal Structure of a Group I Ribozyme Domain: Principles of RNA Packing 著者: Cate, J.H. / Gooding, A.R. / Podell, E. / Zhou, K. / Golden, B.L. / Kundrot, C.E. / Cech, T.R. / A Doudna, J. #3:  ジャーナル: Structure / 年: 1996 ジャーナル: Structure / 年: 1996タイトル: Metal-binding Sites in the Major Groove of a Large Ribozyme Domain 著者: Cate, J.H. / Doudna, J.A. #4:  ジャーナル: Nat.Struct.Biol. / 年: 1997 ジャーナル: Nat.Struct.Biol. / 年: 1997タイトル: A Magnesium Ion Core at the Heart of a Ribozyme Domain 著者: Cate, J.H. / Hanna, R.L. / Doudna, J.A. | ||||||
| 履歴 |
|
- 構造の表示
構造の表示
| 構造ビューア | 分子:  Molmil Molmil Jmol/JSmol Jmol/JSmol |
|---|
- ダウンロードとリンク
ダウンロードとリンク
- ダウンロード
ダウンロード
| PDBx/mmCIF形式 |  1hr2.cif.gz 1hr2.cif.gz | 194.8 KB | 表示 |  PDBx/mmCIF形式 PDBx/mmCIF形式 |
|---|---|---|---|---|
| PDB形式 |  pdb1hr2.ent.gz pdb1hr2.ent.gz | 146.5 KB | 表示 |  PDB形式 PDB形式 |
| PDBx/mmJSON形式 |  1hr2.json.gz 1hr2.json.gz | ツリー表示 |  PDBx/mmJSON形式 PDBx/mmJSON形式 | |
| その他 |  その他のダウンロード その他のダウンロード |
-検証レポート
| 文書・要旨 |  1hr2_validation.pdf.gz 1hr2_validation.pdf.gz | 419.5 KB | 表示 |  wwPDB検証レポート wwPDB検証レポート |
|---|---|---|---|---|
| 文書・詳細版 |  1hr2_full_validation.pdf.gz 1hr2_full_validation.pdf.gz | 438.2 KB | 表示 | |
| XML形式データ |  1hr2_validation.xml.gz 1hr2_validation.xml.gz | 16.5 KB | 表示 | |
| CIF形式データ |  1hr2_validation.cif.gz 1hr2_validation.cif.gz | 25.1 KB | 表示 | |
| アーカイブディレクトリ |  https://data.pdbj.org/pub/pdb/validation_reports/hr/1hr2 https://data.pdbj.org/pub/pdb/validation_reports/hr/1hr2 ftp://data.pdbj.org/pub/pdb/validation_reports/hr/1hr2 ftp://data.pdbj.org/pub/pdb/validation_reports/hr/1hr2 | HTTPS FTP |
-関連構造データ
| 関連構造データ |  1gidS S: 精密化の開始モデル |
|---|---|
| 類似構造データ |
- リンク
リンク
- 集合体
集合体
| 登録構造単位 | 
| ||||||||
|---|---|---|---|---|---|---|---|---|---|
| 1 | 
| ||||||||
| 2 | 
| ||||||||
| 単位格子 |
|
- 要素
要素
| #1: RNA鎖 | 分子量: 51091.352 Da / 分子数: 2 / 変異: DELETION OF C209 / 由来タイプ: 合成 詳細: The RNA was prepared by transcription with T7 RNA polymerase #2: 化合物 | ChemComp-MG / #3: 水 | ChemComp-HOH / | |
|---|
-実験情報
-実験
| 実験 | 手法:  X線回折 / 使用した結晶の数: 1 X線回折 / 使用した結晶の数: 1 |
|---|
- 試料調製
試料調製
| 結晶 | マシュー密度: 3.37 Å3/Da / 溶媒含有率: 63.46 % | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 結晶化 | 温度: 293 K / 手法: 蒸気拡散法, シッティングドロップ法 / pH: 6.5 詳細: MPD, magnesium chloride, sodium cacodylate, spermine, sodium chloride, pH 6.5, VAPOR DIFFUSION, SITTING DROP, temperature 293K | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 |
| ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 結晶化 | *PLUS 温度: 30 ℃詳細: drop is set up using 2:8:5 ratio of solutions a, b and c | ||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||||
| 溶液の組成 | *PLUS
|
-データ収集
| 回折 | 平均測定温度: 100 K |
|---|---|
| 放射光源 | 由来:  シンクロトロン / サイト: シンクロトロン / サイト:  ALS ALS  / ビームライン: 5.0.1 / 波長: 1.1 Å / ビームライン: 5.0.1 / 波長: 1.1 Å |
| 検出器 | タイプ: ADSC QUANTUM 4 / 検出器: CCD / 日付: 1999年3月24日 / 詳細: W16 wiggler |
| 放射 | モノクロメーター: single crystal, cylindrically bent プロトコル: SINGLE WAVELENGTH / 単色(M)・ラウエ(L): M / 散乱光タイプ: x-ray |
| 放射波長 | 波長: 1.1 Å / 相対比: 1 |
| 反射 | 解像度: 2.25→30 Å / Num. all: 64900 / Num. obs: 64900 / % possible obs: 98 % / Observed criterion σ(I): -3 / 冗長度: 4.8 % / Rmerge(I) obs: 0.051 / Net I/σ(I): 22.7 |
| 反射 シェル | 解像度: 2.25→2.33 Å / 冗長度: 4.4 % / Rmerge(I) obs: 0.578 / Mean I/σ(I) obs: 2.4 / Num. unique all: 6463 / Rsym value: 57.8 / % possible all: 99.3 |
| 反射 シェル | *PLUS % possible obs: 99.3 % |
- 解析
解析
| ソフトウェア |
| ||||||||||||||||||||||||||||||||||||||||||
|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|---|
| 精密化 | 構造決定の手法:  分子置換 分子置換開始モデル: PDB entry 1gid 解像度: 2.25→30 Å / Isotropic thermal model: isotropic / 交差検証法: THROUGHOUT / σ(F): 0 / σ(I): 0 立体化学のターゲット値: G. Parkinson, J. Vojtechovsky, L. Clowney, A.T. Brunger, H.M. Berman, New Parameters for the Refinement of Nucleic Acid Containing Structures, Acta Cryst. D, 52, 57-64 (1996). 詳細: used maximum likelihood function as implemented by CNS
| ||||||||||||||||||||||||||||||||||||||||||
| 原子変位パラメータ | Biso mean: 75.4 Å2
| ||||||||||||||||||||||||||||||||||||||||||
| Refine analyze |
| ||||||||||||||||||||||||||||||||||||||||||
| 精密化ステップ | サイクル: LAST / 解像度: 2.25→30 Å
| ||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 |
| ||||||||||||||||||||||||||||||||||||||||||
| LS精密化 シェル |
| ||||||||||||||||||||||||||||||||||||||||||
| ソフトウェア | *PLUS 名称: CNS / 分類: refinement | ||||||||||||||||||||||||||||||||||||||||||
| 拘束条件 | *PLUS
|
 ムービー
ムービー コントローラー
コントローラー








 PDBj
PDBj
































